网络会议预告: Byos对修饰肽段打分及Feature Finder
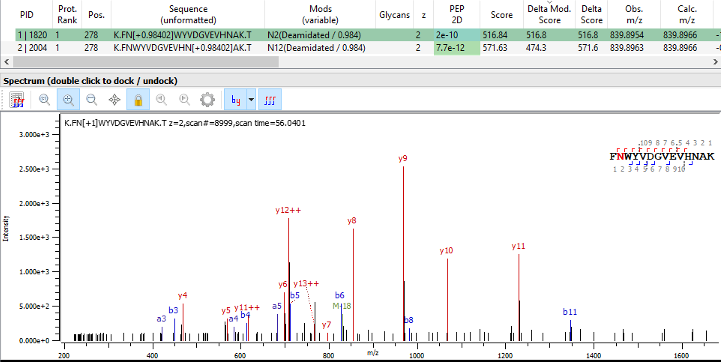
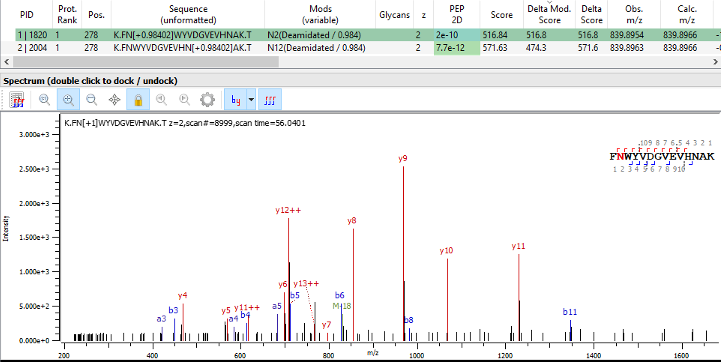
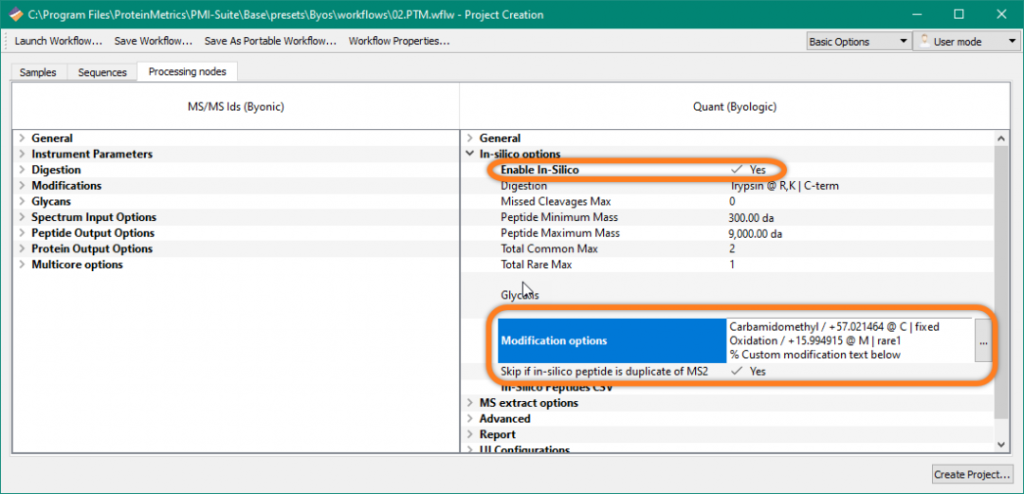
本期Protein Metrics Insights的主要内容: 介绍MS/MS搜索结果中一个重要但鲜为人知的分值Delta Mod score(文末有彩蛋); 展示Feature Finder的新用户界面。 1. 高级功能 Delta Mod Score 当分析翻译后修饰,序列突变,二硫键或其他肽段可变修饰时,关于肽段序列修饰位置的置信度这类有价值的信息记录在Delta Mod. Score值中。Delta Mod. Score的定义是MS/MS图谱中不同修饰匹配分数最高值与次优匹配的差值。在实际应用中,一个肽段有两个可能的修饰位点,Delta Mod. Score分值高或低表示在确定位点的修饰可信度。可信度由两个可能位点得到的MS/MS碎片离子的分布决定。 例如,FNWYVDGVEVHNAK肽段有两个天冬酰胺残基,因此有两个可能的脱酰胺可变修饰位点。在下面的MS/MS ID和谱图中,Byonic 显示脱酰胺修饰位点在第一个天冬酰胺残基上(FN[+1]WYVDGVEVHNAK)。 Delta Mod. Score的值的区间范围为0到肽段的打分值。从结果列表中,我们可以看出Delta Mod. Score等于肽段的打分值,已经达到该值可能的最高值。这表明该修饰位点的可信度非常高。在上面的谱图里可以找到有力的证据表明第一个天冬酰胺脱酰胺化了,而不是第二个天冬酰胺。这是由谱图上两个位点之间的片段佐证,即从y4到y12和b3到b11。 相比之下,下面的例子是一个含有三个天冬酰胺残基的肽段,这里MSMS ID的Delta Mod. Score为0. 尽管第一个天冬酰胺残基和第二个天冬酰胺残基之间有碎片离子,但是第二个天冬酰胺残基和第三个天冬酰胺残基之间没有碎片离子。因此这个修饰位置不可信,脱酰胺化可能发生在第二个天冬酰胺残基和第三个天冬酰胺残基之间,但是谱图中找不出相关证据。值得注意的是:这并不表示是假阳性的ID。这个肽段的脱酰胺化是毫无疑问的,只是不确定肽段序列上哪些位点发生了脱酰胺化。 Delta Mod Score在序列变异分析中是一个非常有用的数值,尤其是对于有多个可能修饰位置的肽段来说尤其重要。在Byos肽谱分析工作流、Byonic Viewer和Byologic项目中都能计算Delta Mod Score。 如果大家想了解更多关于Delta Mod Score干货,请扫描下面二维码报名4月9号(周四)下午2点的网络会议,听Protein Metrics中国技术专家在线解读。报名成功后,会收到Cisco Webinar链接。 2. 新功能展示 V3.7 中Feature Finder的新用户界面 最近发布的V3.7版本中更新了肽谱分析中Feature Finder算法的使用界面。 Feature Finder扫描一个样品中所有可能的肽段信号的MS1信息。使用Feature Finder能够确保所有峰都会监测到,包括没有被MS/MS鉴定到的峰和in-silico peptides方法鉴定到的峰。Feature …