
ADME Suite Overview
ADME性质的计算和预测
计算模拟预测目标物的吸收,分布,代谢和排泄(ADME)特性,支持对库的高通量筛选,提供对药理作用的深入了解,有助于确保产品对人类的使用是安全的.
ADME Suite可提供基于结构的高质量药物动力学特性计算。
- 基于结构预测
- 血脑屏障透过性
- 细胞色素P450*酶系的抑制性和底物特异性
- 分布*
- 临床实验推荐日最大剂量
- 口服生物利用度-基于logP以及剂量
- 被动吸收
- P-糖蛋白特异性结合
- 理化性质- logP,* logD,* pKa,* 水溶性,* 等.
- 代谢位点
- 提供预测置信度评估
- 搜索对应模型的内部数据库的实验数据
- 用实验数据训练预测模型,以更好地反映新的化学空间
- 包含自定义模型和自定义预测算法
*表示这些模型支持自添加数据训练
Benefits
全面的ADME性质计算
易于使用
- 只需绘制您的结构即可进行预测
- 无需成为软件工程师,程序员或计算化学家即可训练模型

快速精准和可靠
- 快速计算单个化合物或数万个化合物的性质
- 基于精心挑选的实验数据进行预测
- 通过可靠性指标,数据库中类似结构的显示以及原始实验数据的文献参考轻松衡量结果质量
方便的可视化
- 通过对结构进行颜色映射,可视化子结构/原子对属性值的贡献(某些模块)
- 通过用户定义的电子表格结果颜色编码,快速识别库中的有利和不利化合物

深入理解数据结果
- 通过创建散点图,浏览,过滤,排序和排序库的工具轻松识别趋势并确定化合物的优先级
- 集中掌握每个分子的完整特性,自信地做出决定

可自定义使用内部数据
- 从商业产品获得内部模型的准确性。 使用您自己的实验数据来扩展可训练模块的适用范围。

可扩展到第三方模型
- 通过包含第三方和内部模型来创建用于预测数据的单一环境
1绘制/导入结构
2选择感兴趣的属性
3查看结果并做出决定
4方便地报告为PDF或复制/粘贴
Product Features
ADME计算功能特点
一般特征
基于属性的结构修改和线索优化
血脑屏障通透性
细胞色素P450抑制剂
细胞色素P450底物
分配
推荐的最大每日剂量
口服生物利用度
被动吸收
P-gp特异性
PK探索
代谢区域选择性
一般特征
- 基于结构计算有机分子的ADME属性(可在软件内绘图,或从第三方绘图软件复制/粘贴); SMILES字符串输入; InChI代码; 导入MOL,SK2,SKC或CDX文件输入; 或在内置字典中按名称搜索
- 自动检测互变异构形式(适用于预测模块)
- 选择规范或主要形式
- 高亮显示结构以指示子结构/原子贡献(某些模块)
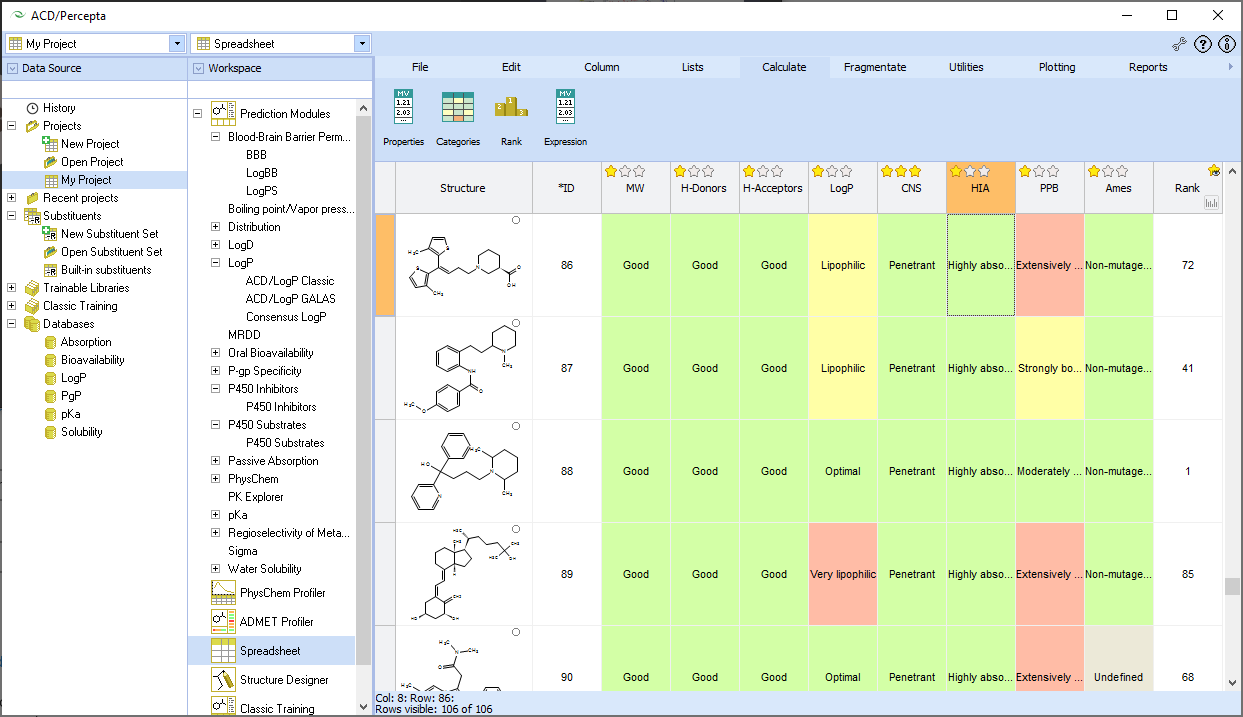
- 批量计算化合物组或库的属性,并使用内置工具对结果进行排序,过滤,绘图和排名
- 设置用户定义的标签颜色
- 过滤结果
- 按升序/降序对结果进行排序
- 在历史记录中检索先前计算的值的结果
- 将结果报告为PDF或复制/粘贴到您选择的应用程序中
- 在以下模块中使用自有实验数据训练— CYP450抑制,分布,LogP,LogD,pKa和P-gp特异性
- 通过使用XML协议或DLL的形式连接到现有的Web服务来添加自定义模型/算法和内部预测算法(仅在Web Portal部署中可用)
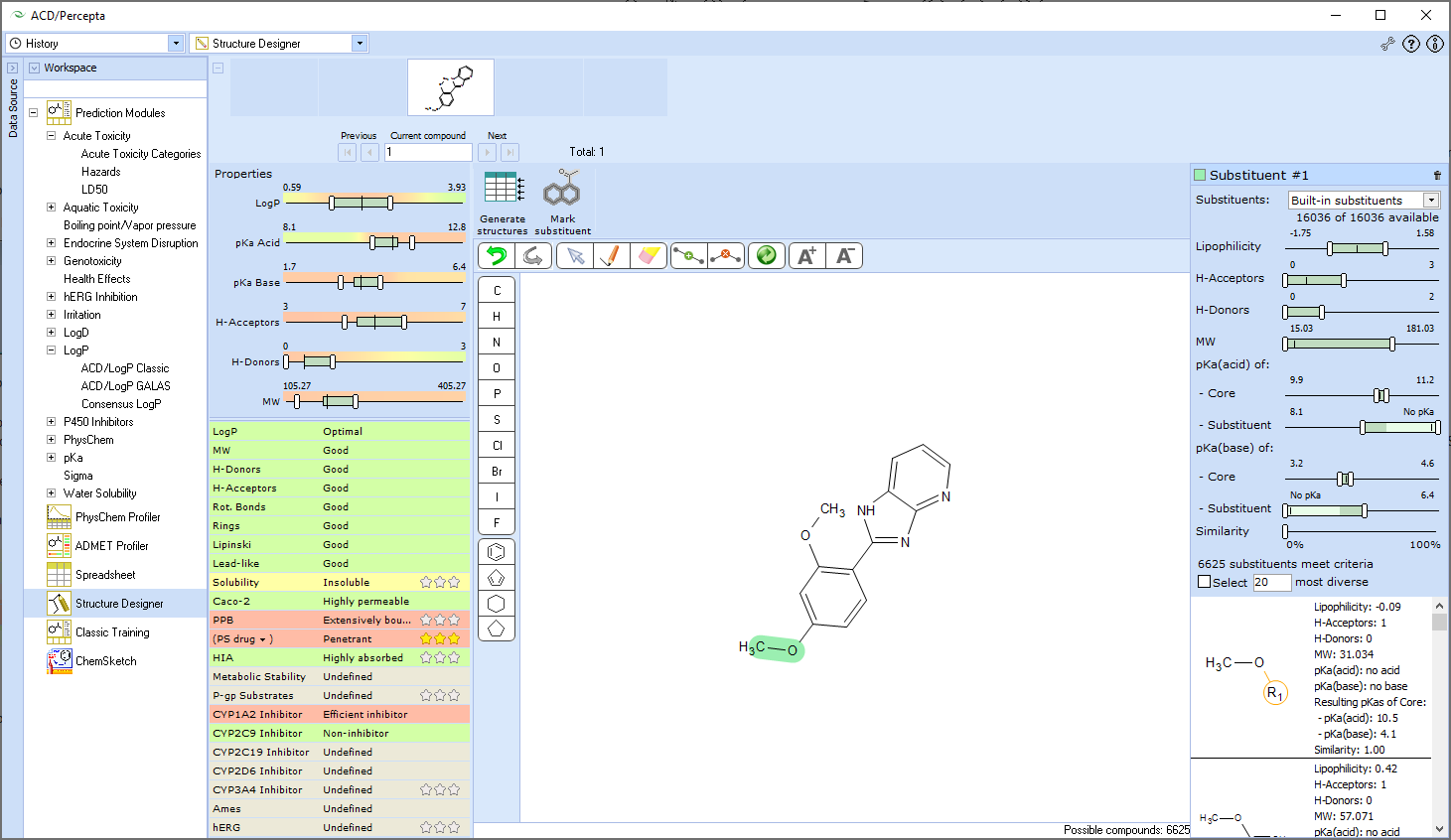
基于属性的结构修改和线索优化
- 深入了解结构属性关系
- 了解并修改化合物的药代动力学特征
- Lipinski的5法则,先导物法则,细胞通透性,血浆蛋白结合,CNS渗透,代谢稳定性和CYP抑制的指标
- 使用交互式优化工具修改结构
- 根据最佳特性生成具有取代基修饰的类似物库
- 根据您所需的特性对数百个结构类似物进行排序,过滤和确定优先级
- 创建和使用自定义片段库
- 使用内置的逆合成工具靶向可合成的片段
血脑屏障通透性
- 定量预测血脑屏障(BBB)的渗透性。
- 脑渗透率(logOS)
- 脑渗透的程度(logBB)
- 脑/血浆平衡率(log(PS * fu, brain))
- 改变基本理化特性的值(logP,pKa,血浆中未结合分数),以研究对血脑屏障通透性的影响
- 结果可视化
- 渗透性的“交通灯”指示器
- 用著名的CNS活性和外周活性药物研究的化合物的BBB渗透图
- 提示化合物在BBB中易于扩散或主动外排
- LogPS和LogBB的子模块提供详细的预测结果
- 内部库中最多提供3种最相似的结构,并提供实验值和文献参考
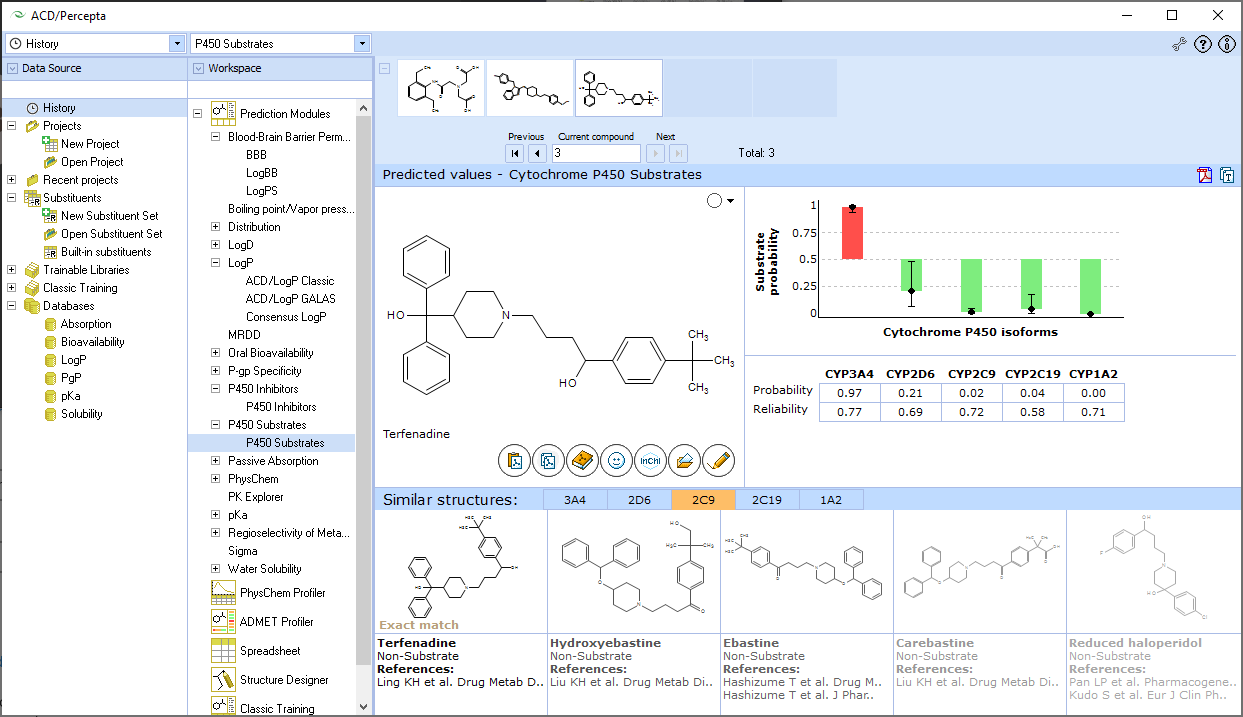
细胞色素P450抑制剂
- 计算化合物对5种主要人类细胞色素P450亚型表现出“一般”(IC50 <50μM)或“有效”(IC50 <10μM)抑制的可能性:3A4、2D6、2C9、2C19、1A2
- 结果显示为带有置信区间的条形图
- 估计每个结果的预测准确性
- 提供可靠性指数,并在内部库中显示5个最相似的结构,并提供实验值和文献参考
- 可用实验数据训练算法
细胞色素P450底物
- 计算化合物被细胞色素P450(异构体3A4、2D6、2C9、2C19、1A2)代谢的可能性
- 结果显示为带有置信区间的条形图
- 估计每个结果的预测准确性
- 可靠性指数,并在内部库中显示5个最相似的结构,并提供实验值和文献参考
- 用实验数据训练算法
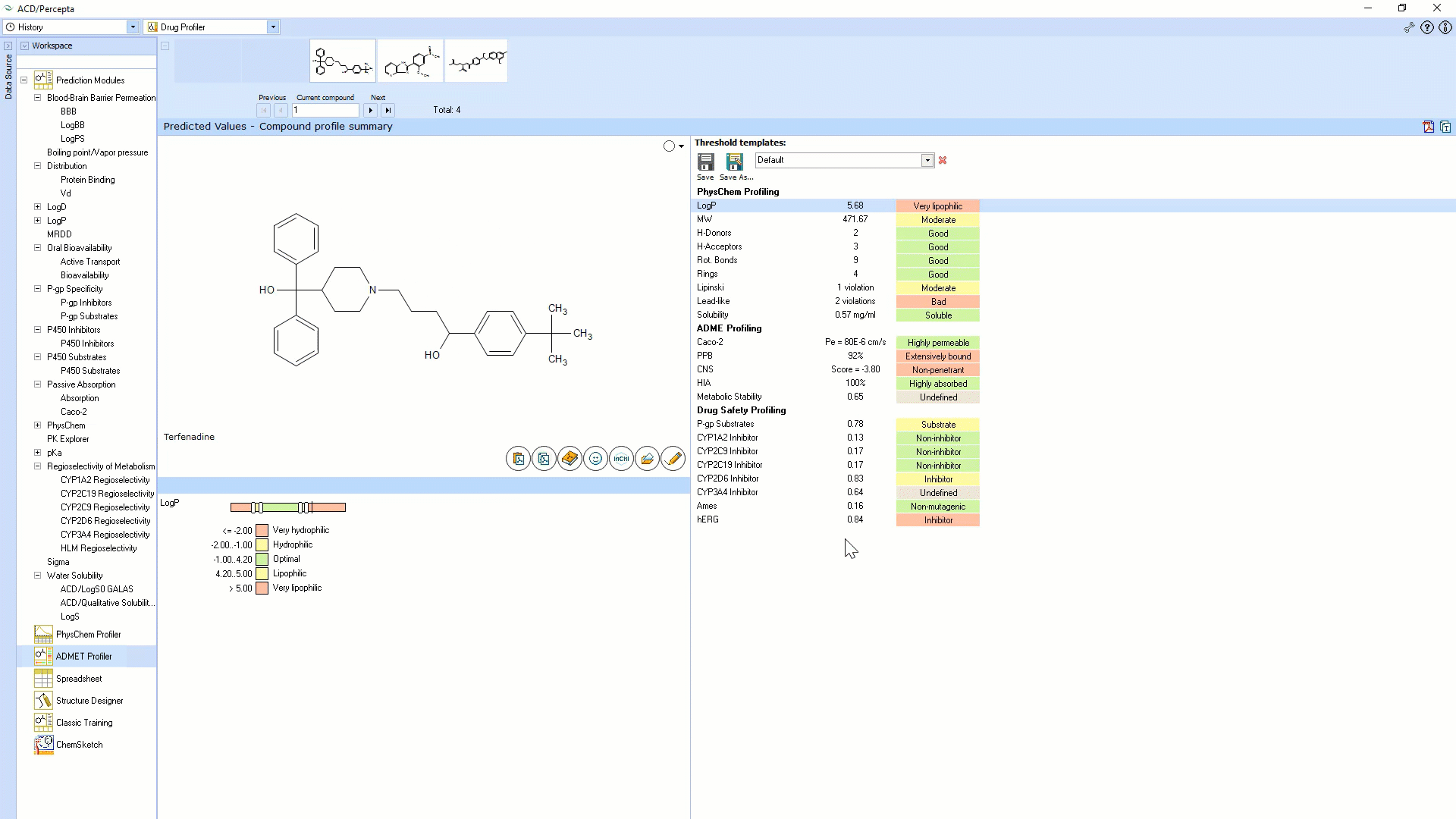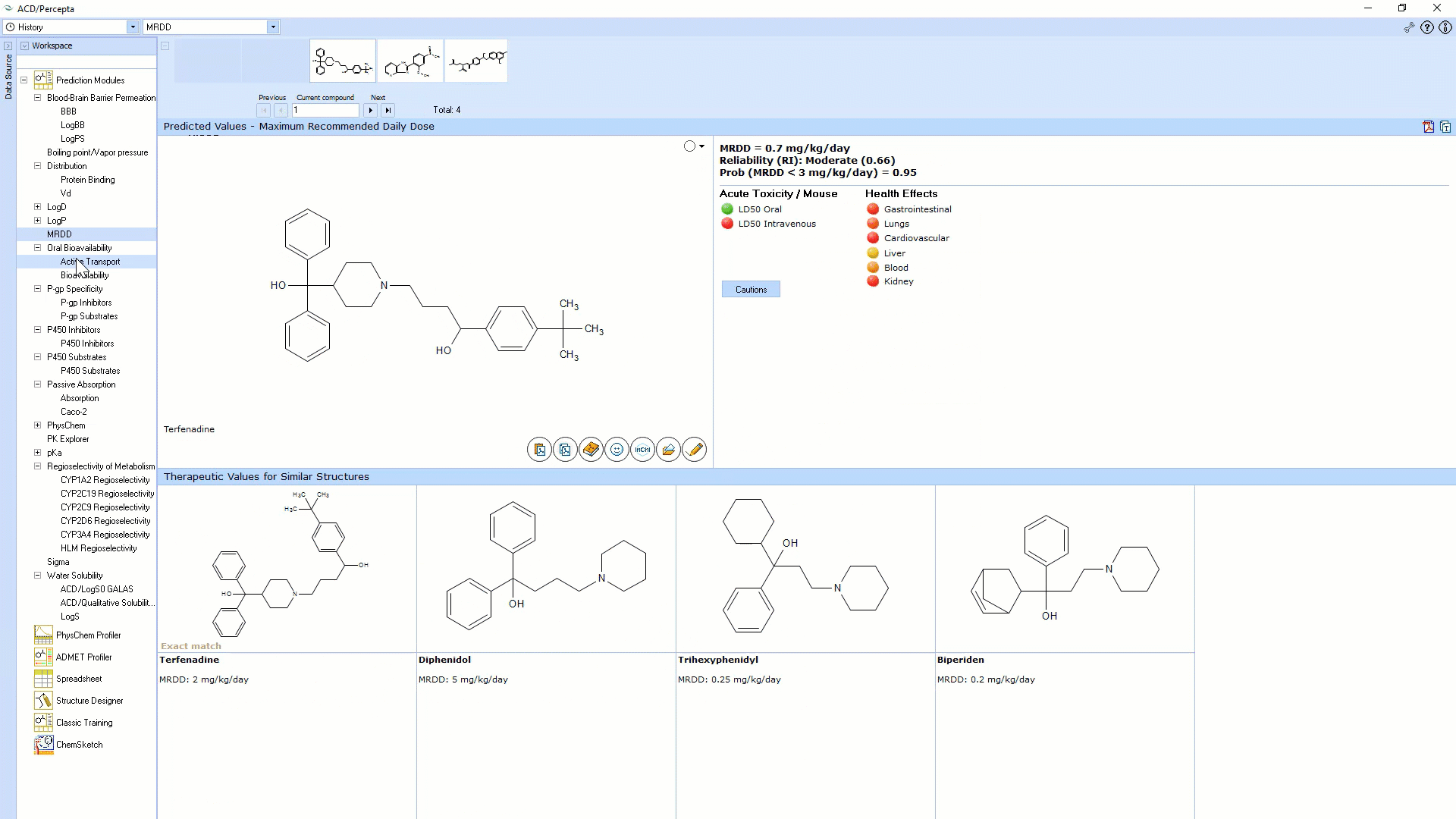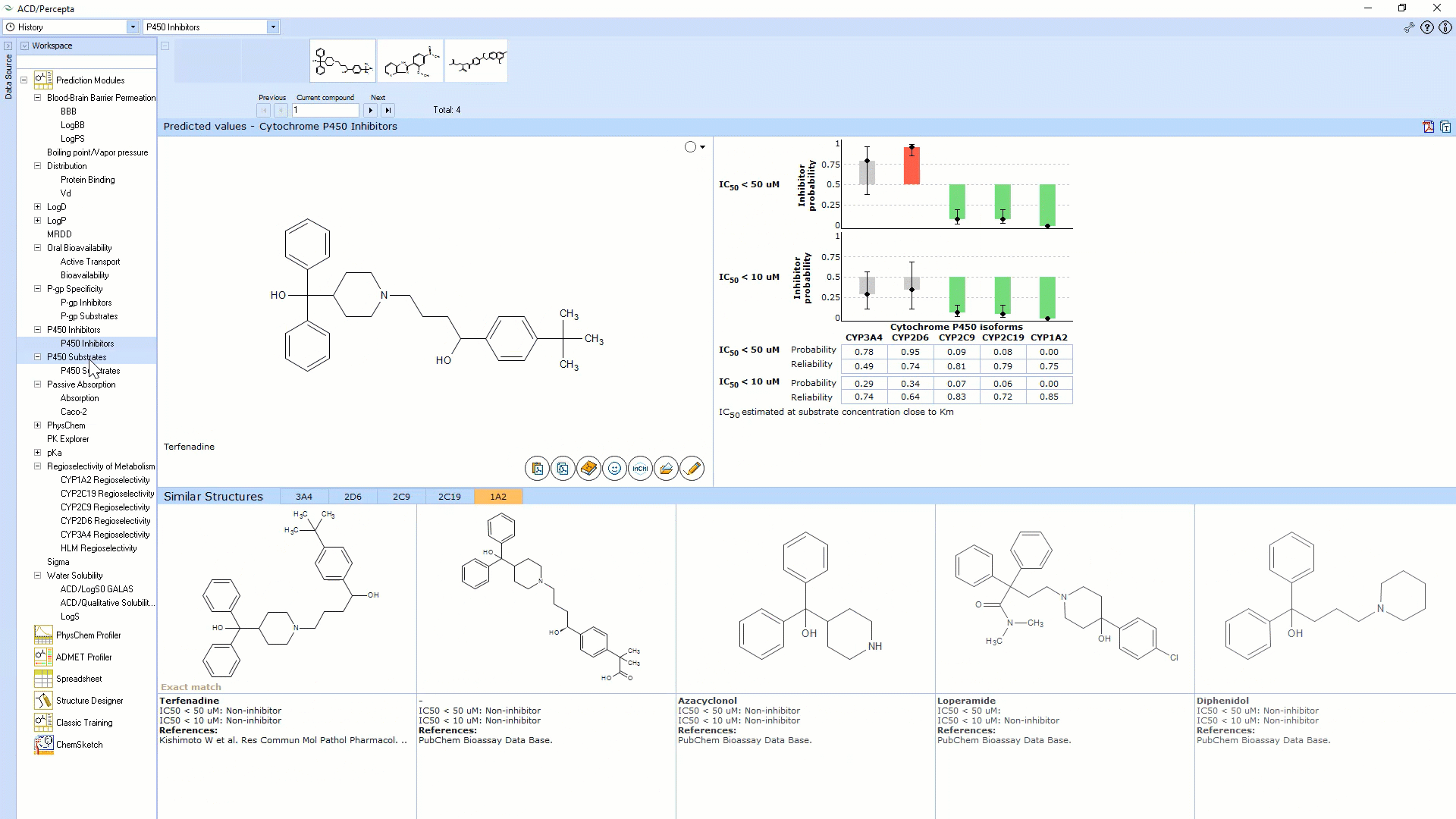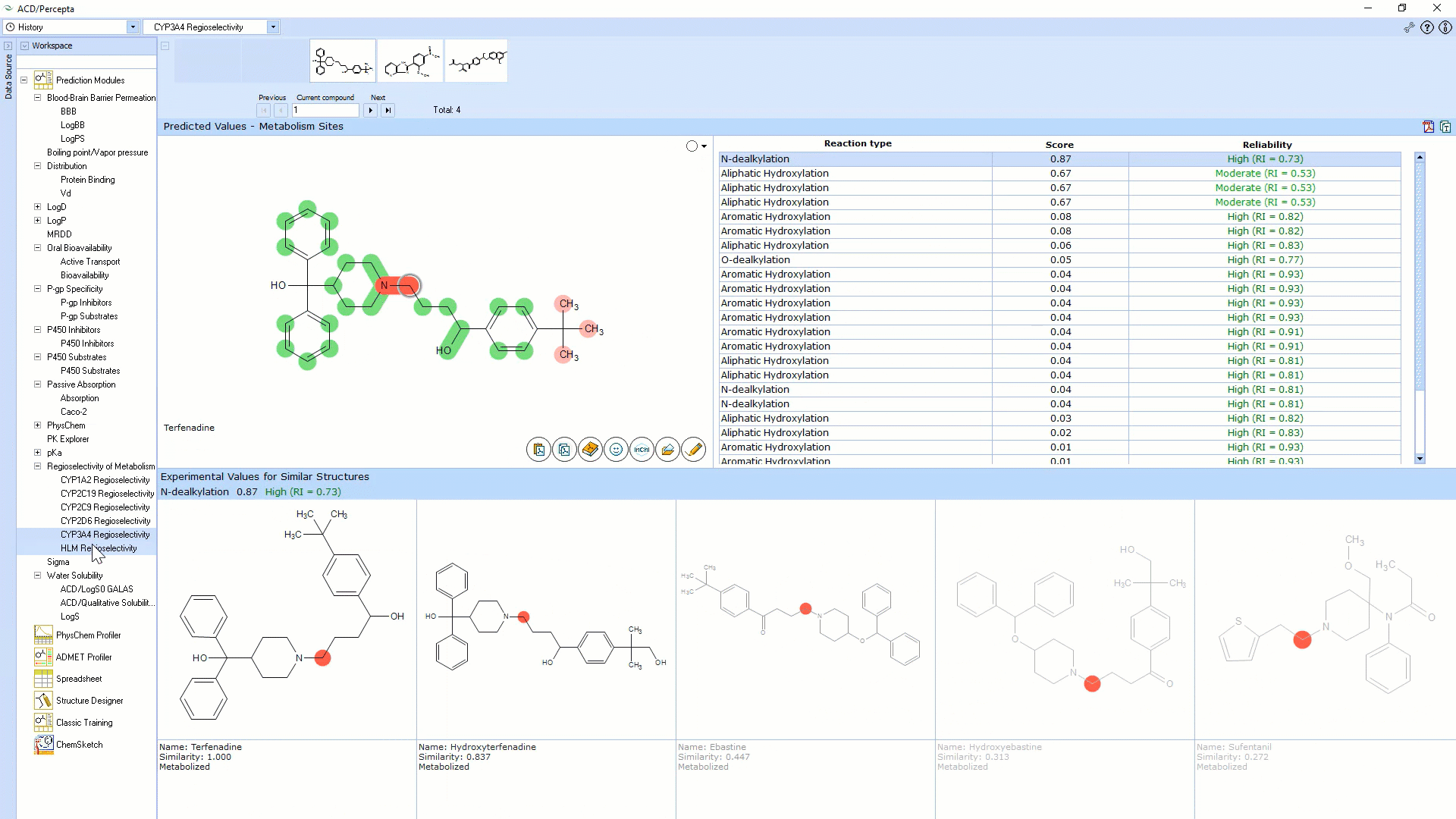
分配
血浆蛋白结合
- 预测血浆蛋白结合(PPB)
- 与人血浆蛋白结合的百分比(%PPB)
- 对血清白蛋白的亲和力常数(logKaHSA)
- 有助于结合的血浆蛋白列表
- 预测准确性的估计
- 可靠性指数,并在内部库中显示5个最相似的结构,并提供实验值和文献参考
- 训练实验数据
分布体积
- 根据生理学Øie-Tozer模型计算组织结合的程度和分布体积(Vd)
- 更改关键基础属性的值(logP和血浆中的未结合分数),以研究对分布到组织中的影响
- 预测准确性的估计
- 在内部库中显示5个最相似的结构,并提供实验值和参考文献
推荐的最大每日剂量
- 计算人类建议的口服最大每日剂量(mg / kg /天)
- 预测准确性的估计
- 可靠性指数,并在内部库中显示5种最相似的药物,并提供实验值和文献参考
- 指示对器官的不良影响
- 指示小鼠毒性(口服和静脉内给药)
- 对不可靠的预测发出警报
口服生物利用度
主动运输
- 预测化合物是否可能是寡肽转运蛋白(PepT1)或胆汁酸转运蛋白(ASBT)的底物
- 预测由其他系统进行的载体介导的运输:单羧酸转运蛋白MCT1,氨基酸载体等。
- 类似结构的参考数据
生物利用度
- 预测口服后达到全身循环的药物百分比(%F)
- 探索剂量依赖性
- 查看影响口服生物利用度的终点的贡献:溶解度,稳定性(pH <2),被动吸收,首过代谢,P-gp外排,主动转运
- 用颜色编码显示的结果表明生物利用度良好或不良
- 预测准确性的估计
- 在内部库中最多显示5个相似结构,并提供实验值和参考文献
被动吸收
吸收性
- 基于logP和pKa预测空肠上皮的被动通透性
- 可输入实验测量值以改善预测
- 改变logP和pKa值以模拟亲脂性和离子化对肠渗透率的限制作用
- 计算口服吸收的程度(%HIA)
- 估计跨细胞和旁细胞途径对总%HIA的相对贡献
- 提供对Absorption DB的访问权限-完全可浏览和搜索的数据库,其中包含用于开发HIA模型的实验数据以及相应的文献参考
- 展示内部数据中3个相似结构的相关属性的实验值
Caco-2
- 根据logP和pKa值或特定pH下的logD,在指定pH和搅拌条件下计算Caco-2细胞单层的被动渗透性
- 调整pH值和搅拌速度
- 改变logD或logP和pKa值以模拟亲脂性和电离对Caco-2渗透速率的限制作用
- 有关跨细胞和旁细胞途径对总体Caco-2渗透性的相对贡献的信息
- 预测准确性的估计
- 在内部库中最多显示3个相似的结构,并提供实验信息和参考文献
P-gp特异性
P-gp抑制剂
- 估计化合物抑制P-糖蛋白的可能性
- 根据结构特征和理化性质将化合物分类为P-糖蛋白的抑制剂或非抑制剂
- 预测准确性的评估
- 具有实验值和参考文献的内部库中5个最相似结构的预测和显示的可靠性
- 训练实验数据
P-gp底物
- 估计化合物是P-糖蛋白底物和高亲和力底物的可能性
- 根据电离,分子大小和化合物类别(肽,生物碱,蒽环类等),将化合物分类为P-糖蛋白的底物或非底物
- 预测准确性的估计
- 具有实验值和参考文献的内部库中5个最相似结构的预测和显示的可靠性
- 训练实验数据
PK探索
探索各种动力学参数对理化性质的依赖性
- 可视化以下参数对剂量,logP和pKa的依赖性(结果以图形图形式提供。使用预测值或为logP和pKa输入实验得出的值):
- %F-LogP(确定剂量下口服生物利用度对logP的依赖性)
- Cp(Max)-LogP(在定义的剂量下,最大可达到的药物浓度对logP的依赖性)
- %F-剂量(生物利用度-剂量关系)
- Cp(Max)-剂量(不同剂量下可达到的最大药物血浆水平)
- Cp-时间(口服和静脉内给药的血浆浓度-时间曲线模拟)
- 根据输入的理化特性值,估算吸收率(ka),全身清除率(ke),胃肠道溶解度(SolGI),分布体积(Vd)和肠道和肝脏的系统前代谢(首过清除率)
- 更改这些值中的任何一个以重新计算
- 选择忽略首过效应
- 计算口服和静脉给药后可达到的最大血浆水平(Cp(Max))和相应的时间(Tmax),浓度-时间曲线下的面积(AUC),以及口服生物利用度(%F)
代谢区域选择性
- 预测人肝微粒体(HLM)和五种主要的细胞色素P450酶(1A2、2C19、2C9、2D6、3A4)代谢的代谢位点,以帮助:
- 识别新化学实体上的代谢位点
- 指导具有改善代谢特性的化合物的合成
- 识别并阐明可能的代谢物结构
- 结构上的颜色映射表示每个原子处新陈代谢的可能性
- 每个原子作为新陈代谢的可能部位的概率得分(0-1)
- 查看每个原子上预期的代谢反应类型:脂族或芳族羟基化,N-脱烷基,O-脱烷基或S-氧化
- 预测准确性的估计
- 自内部库中提取5个最相似结构的预测和显示可靠性。 指示代谢部位和实验结果的相似性(代谢或未代谢)
Deployment/Integration Options
选择适合您的部署选项

桌面版
在单独的计算机上安装ACD / ADME Suite,以使用完整的图形用户界面并使用您自己的数据训练可训练模块。

批量版
用最少的用户干预即可计算成千上万种化合物的ADME性质。 批处理部署与Microsoft Windows和Linux操作系统兼容。可嵌入到公司的工作流工具,例如Pipeline Pilot。

Percepta Portal网页版
使用基于浏览器的应用程序来预测ADME属性。 可以使用KNIME集成组件。 托管在您的公司内网或云上。 适用于Linux和Windows OS。
研发组织经常使用内部收集的实验数据来构建自己的ADME属性预测模型。 尽管这些模型由于代表了所需的化学空间而提供了出色的准确性,但更新和支持内部模型非常耗时。
ADME Suite允许您使用精选的实验数据来扩展训练数据库和可训练模型的适用范围。 因此,在商业产品的支持和广泛适用性的帮助下,您将获得内部模型的准确性。 而且由于培训容易进行,因此您不需要计算化学家或软件工程师。
联系我们咨询产品
填写表单,我们会尽快与您联系
关注我们的微信公众号
软件相关资源,月刊,真实案例,
软件使用小技巧尽收其中!
