环肽定性的难点是缺乏固有的N/C-端和多样化的环化类型,如何在fasta文件中定义环肽的起止残基是搜库成功的关键。Byonic的策略是根据肽键方向(即结构示意图中箭头方向)来线性化环肽,见如下示例:
① 二硫键环化
Somatostatin是通过二硫键(即结构示意图中的曲线)连接的环肽,Byonic将二硫键定义为修饰,可产生1个线性肽,fasta文件如图2所示:
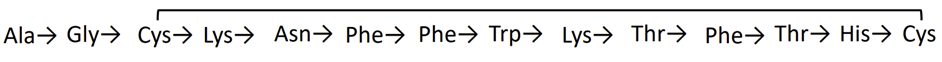
图 1 Somatostatin结构示意图

图 2 Somatostatin线性化序列
② 头尾酰胺化
Unguisin A为环七肽,在每个酰胺键处随机开环,可产生 7 个线性肽;GABA为非标准氨基酸,Byonic可用残基J(精确分子量为100)添加一个固定修饰替代GABA,fasta文件如图4所示:
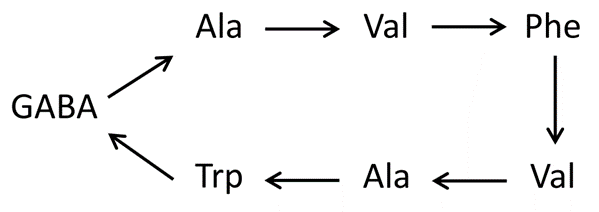
图 3 Unguisin A结构示意图
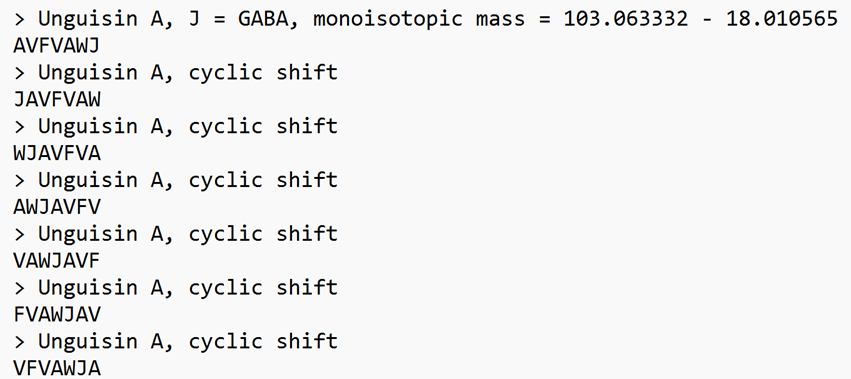
图 4 Unguisin A线性化序列
③ 侧链氨基到C端羧基酰胺化
Polymyxin B1含有大量非标准氨基酸Dab(2,4-二氨基丁酸)和1个脂肪酸,Byonic分别用X和J替代Dab残基和脂肪酸残基,fasta文件如图6所示:
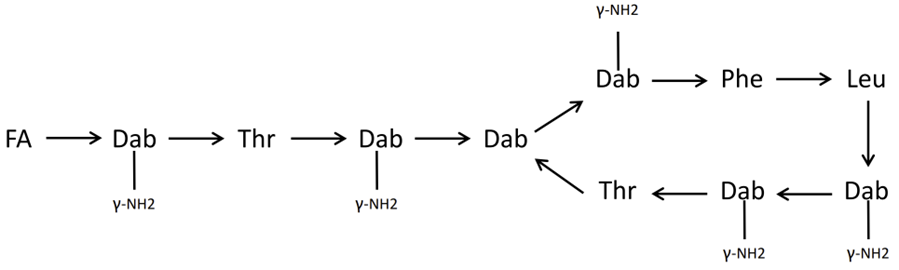
图 5 Polymyxin B1结构示意图

图 6 Poymyxin B1线性化序列
④ 侧链羧基到N端氨基酰胺化
Siamycin II是含有2条二硫键的线性化侧链环肽,根据肽键方向,可产生1个线性肽,fasta文件如图8所示:
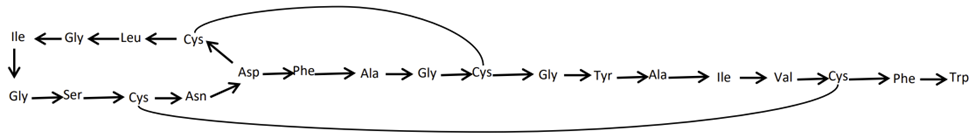
图 7 Siamycin II结构示意图

图 8 Siamycin II线性化序列
去掉Add decoys,否则随着环肽复杂性的提高,一些正确的PSMs也会被过滤掉,如图9所示:
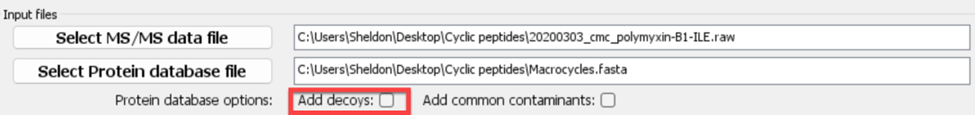
图 9 Add decoys参数设置
酶切位点设为空白,支持环肽Top-down分析,如下图所示:
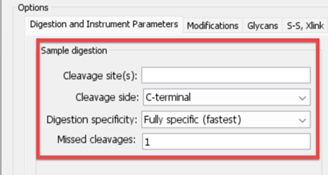
图 10 酶切位点参数设置
① 二硫键环化:在S-S,Xlink选项下,勾上Disulfides即可;如果氨基酸通过交联试剂偶联,可勾上Crosslink:Custom,按格式输入偶联delta mass和偶联位点即可,如图11所示:
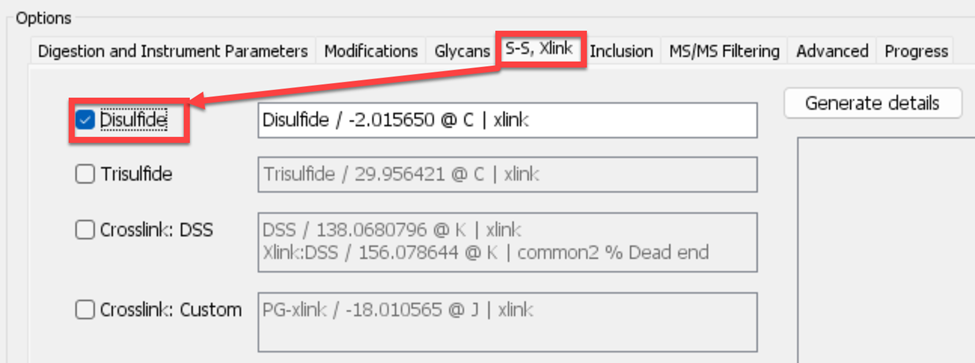
图 11 二硫键环化修饰参数设置
② 头尾酰胺化:Unguisin A含有一个非标准氨基酸GABA,需设为固定修饰;在环肽C端设置脱水的可变修饰,因为环肽可能在体外线性化。这一设置允许Byos搜索所有可能的线性化肽,如图12所示:
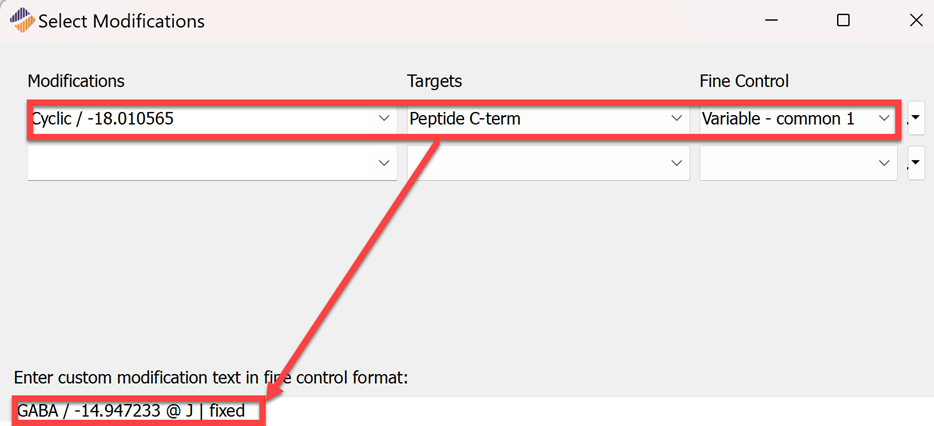
图 12 首尾酰胺化修饰参数设置
③ 侧链氨基到C端羧基酰胺化:Polymyxin B1含有FA和Dab 2个非标准氨基酸,分别用J和X代替,并设为固定修饰;启动wildcard search,搜索可能存在的未知修饰,如图13所示:
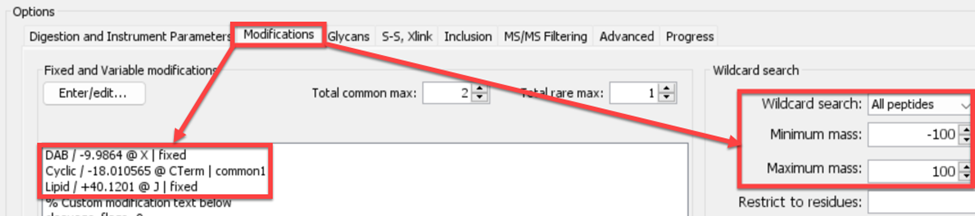
图 13 侧链氨基到C端羧基酰胺化修饰参数设置
④ 侧链羧基到N端氨基酰胺化:Byonic默认二硫键为rare修饰,Siamycin II含有2个二硫键,需将total rare max改为2,如图14所示:
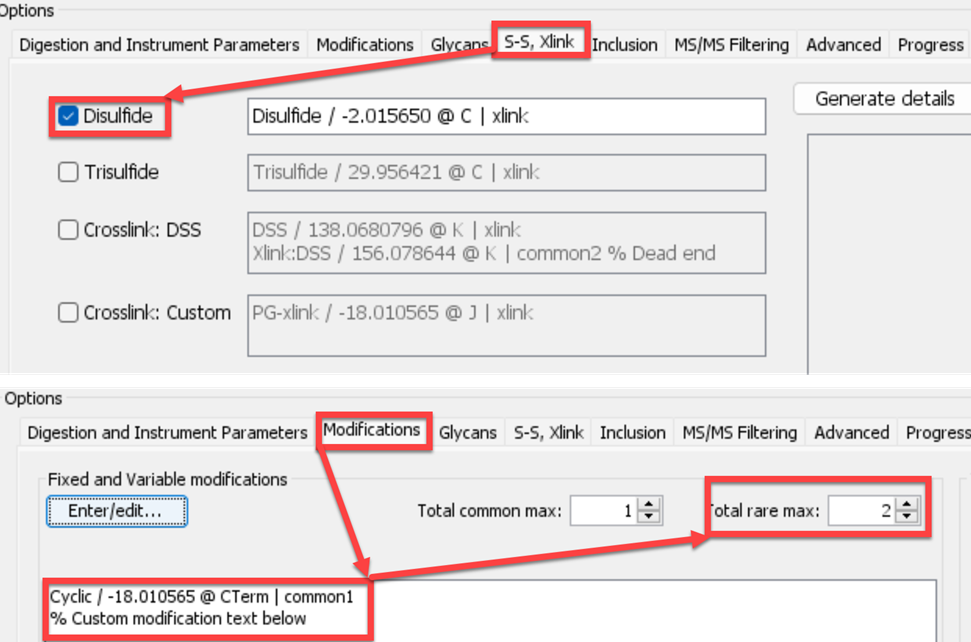
图 14 侧链羧基到N端氨基酰胺化修饰参数设置




