
简介
-
糖对于许多生物过程至关重要,比如蛋白质折叠和细胞间通讯
-
约一半的哺乳动物蛋白质是糖基化蛋白
-
质谱在蛋白质和糖蛋白研究中的应用十分广泛
-
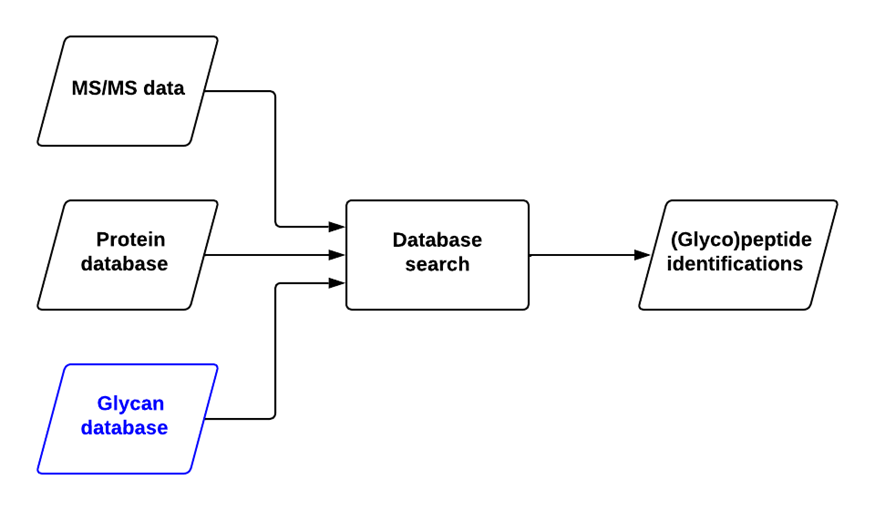
数据库搜索软件是根据串联质谱数据鉴定蛋白质的主要方法。该方法的要求如下:
✔ 完整的蛋白质数据库——得益于方便的基因组测序,此要求通常都可以满足
⚠️ 对于糖蛋白,还需要完整的糖数据库——此要求通常难以满足;往往只具备不完善的信息
-
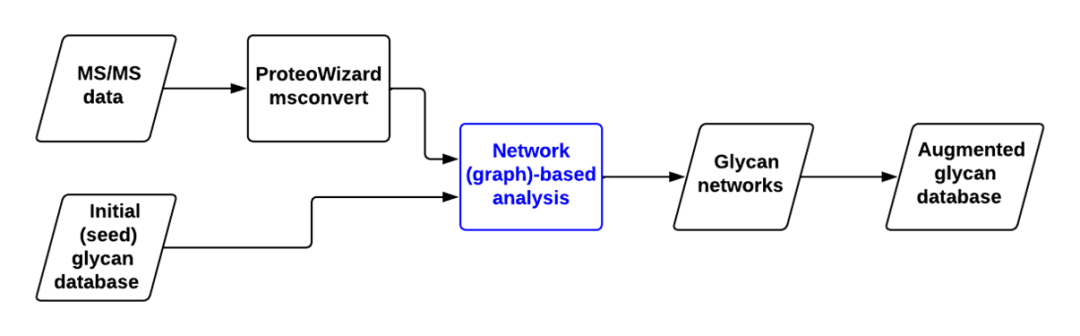
目标是开发出能建立更优的 N-糖数据库的软件,从而根据质谱数据本身建立样品特异性糖数据库,而不是仅仅依赖已有的糖数据库

编写基于网络的分析软件,使用更多糖数据扩充初始(“种子”)糖数据库,从而构建更完整的糖数据库
我们测试过的算法包括:
-
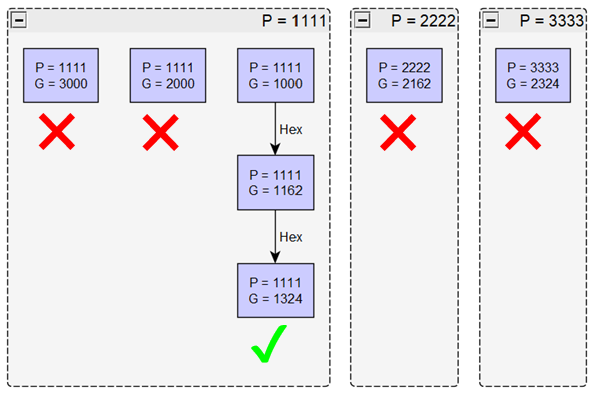
算法 1(不使用网络):对于每一幅 MS/MS 谱图,一旦发现 N-糖基化特征峰就推断出糖并添加到糖列表中
-
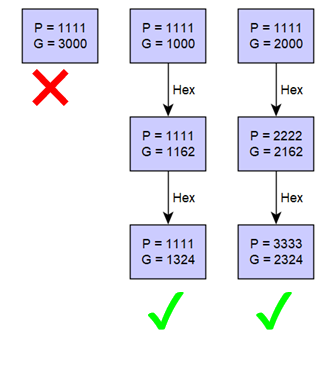
算法 2(单网络):根据算法 1 推断出的糖构建网络或图表,其中每个节点都是一种糖,每条边缘连接质量数相差一个单糖(HexNAc、Hex、Fuc 等)的两个节点。仅从尺寸 ≥ 3 的簇中选取节点构建糖列表。
-
算法 3(多网络):将算法1推断出的糖分成一个个小框,使每个框中的所有糖都具有相同的裸肽质量数。在每个框中,遵循算法 2 的步骤计算。
测试使用的所有数据集均下载自 MassIVE 或 PRIDE
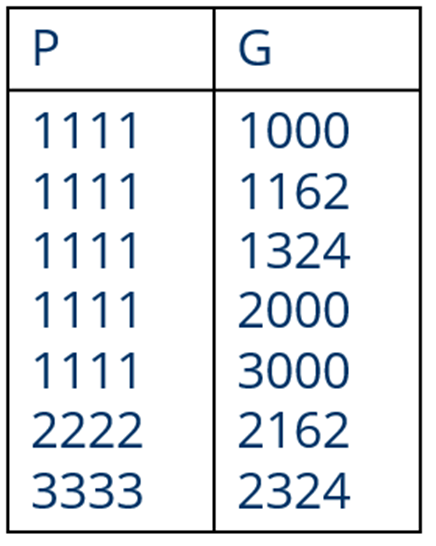
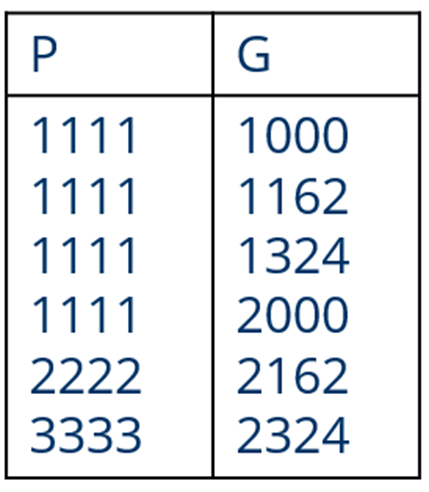
算法 1(不使用网络)
为方便起见,质量数都使用近似整数
P = 裸肽质量数(“裸”= 无糖)
G = 糖质量数
Hex 残基质量数 ≈ 162
算法2和3只接受尺寸≥ 3的簇
算法 2(单网络)
算法 3(多网络)
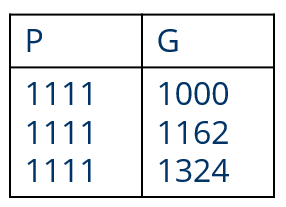
使用不含 N-糖基化结构的样品数据作为阴性对照来测试软件。因此,输出应不含糖。我们用 PNGase F 处理了这些样品,释放出所有 N-糖 (注意:如果酶促反应进行得不完全,可能会残留一些 N-糖)。
输出的糖数量
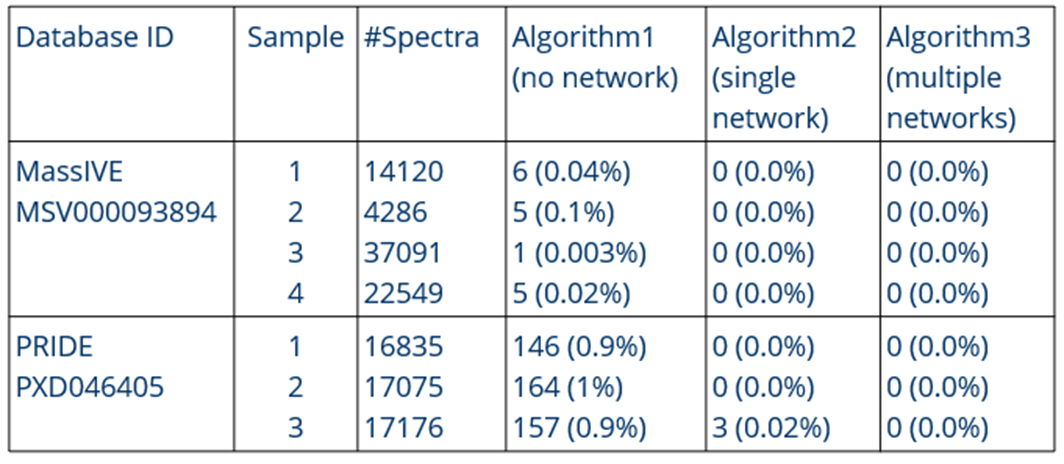
算法 3 产生的假阳性结果最少à用于全部后续工作
-
MassIVE 中的数据集 ID 为 MSV0000088754。出版物 [参考文献 3] 中也有游离糖 MALDI 实验的结果
-
样品来自分别感染了以下两种根瘤菌的大豆 (Glycine max) 根瘤:
-
有固氮能力的野生型 (WT) 慢生根瘤菌 (Bradyrhizobium)
-
无固氮能力的突变型 (M) 慢生根瘤菌 (Bradyrhizobium)
-
我们以 Byonic 数据库“N-glycan 52 plants”作为“种子”数据库开展再分析(原始出版物也使用该数据库作为基础)
-
使用算法 3 进行再分析后,发现了另外 9 种糖
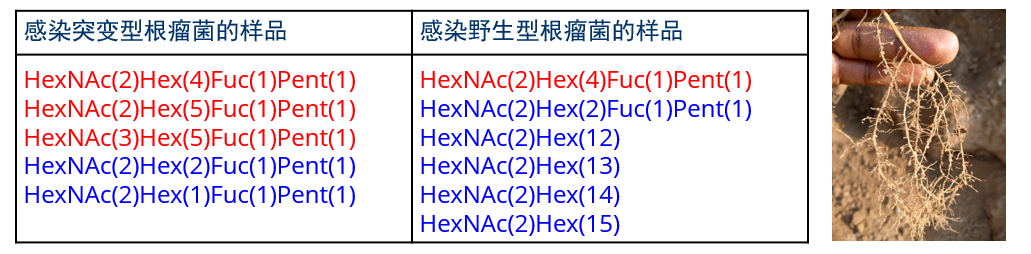
红色:再分析和游离糖 MALDI 实验中都观察到的糖
蓝色:仅在再分析中观察到的糖
-
PRIDE 中的数据集 ID 为 PXD010308。出版物 [参考文献 4] 中也有游离糖 MALDI 实验的结果
-
样品是来自草鱼 (Ctenopharynodonidella idella) 的 IgM
-
我们以 Byonic 数据库“N-glycan 182 human no multiple fucose”作为“种子”数据库开展再分析(原始出版物也使用了该数据库)
-
使用算法 3 进行再分析后,发现了另外 18 种糖

-
通过在扩充的糖数据库中运行数据库搜索 (Byonic),可以(尽可能地)验证我们在再分析中发现的糖
-
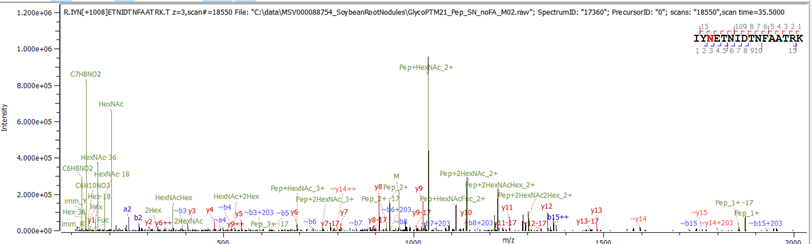
谱图中可标记的高峰越多,糖肽鉴定结果就越可靠
-
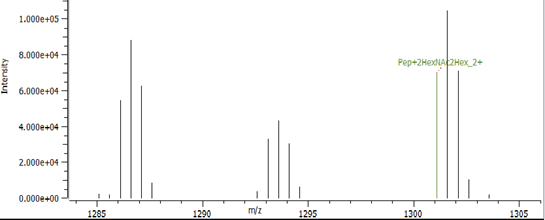
此处的示例显示了来自大豆根瘤 (M) 的 HexNAc(2)Hex(2)Fuc(1)Pent(1)
-
放大视图显示了戊糖峰
-
PRIDE 中的数据集 ID 为 PXD041208
-
样品是由转基因丝状真菌表达系统 Thermothelomyces heterothallica (C1) 产生的单克隆 IgG1 抗体
-
原始出版物 [参考文献 5] 的数据分析中包括一系列高甘露糖 ,即 HexNAc(2)Hex(n),1 ≤ n ≤ 11,以及 HexNAc(3)Hex(n),2 ≤ n ≤ 6
-
使用算法 3 进行再分析后,发现了另外 6 种糖

-
我们还深入分析了这些额外发现的糖的 MS/MS 数据
-
m/z 773 处无峰 à 表明第 3 个 HexNAc 不是平分型 GlcNAc
-
与 HexNAc(2)Hex(…) 相比,HexNAc(3)Hex(…) 中的 Hex 峰(m/z 163、145、127 处)明显更小 à 表明第 3 个 HexNAc 位于末端,且不在 LacNAc 单元中
-
推测:或许 HexNAc(3)Hex(…) 更合适描述为末端带 GlcNAc 的高甘露糖,而不是杂合 N-糖

-
我们进行再分析时得出了另一个有趣的结果,即存在一个额外的大糖“簇”,簇中的每个元素都比已知糖重(几乎恰好)28 Da
-
推测:甲酰化产生的artifact [参考文献 6]

结论
-
从实践角度来看,数据库搜索可以说是根据 MS/MS 数据鉴定蛋白质或糖蛋白最有效的软件
-
但是,数据库搜索鉴定不了库中未收录的蛋白质或糖。本项目旨在通过完善糖数据库来消除或减少“盲点”
-
网络的使用对于该软件的有效性至关重要
-
网络模型模拟了体内糖合成过程
-
我们的网络算法从之前已经发表的数据中发现了更多糖
-
后续通过数据库搜索做了验证
-
与实验结果一致(游离糖 MALDI)

参考文献
作者在此声明,本文不涉及任何竞争性经济利益。
关于Protein Metrics
Protein Metrics LLC是一家全球领先的质谱数据解析软件供应商,公司总部位于美国加州。我们为科研和企业用户提供高效准确的一站式质谱数据解析方案,帮助用户发现、解决问题。Protein Metrics在全球范围内提供销售和支持,目前已为超过150个企业和300个科研单位提供服务。
联系我们邀约演示:
王蕾 13482181958



