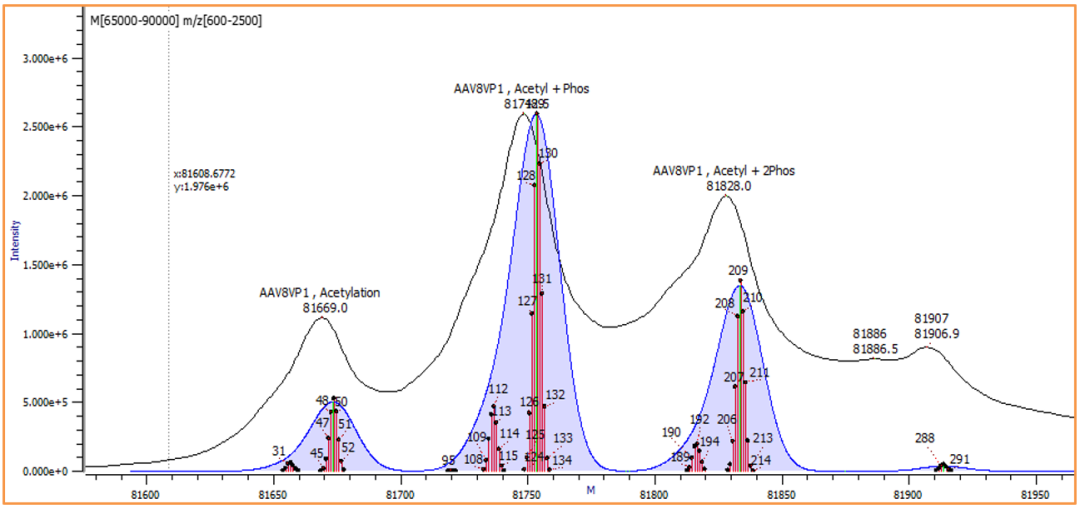
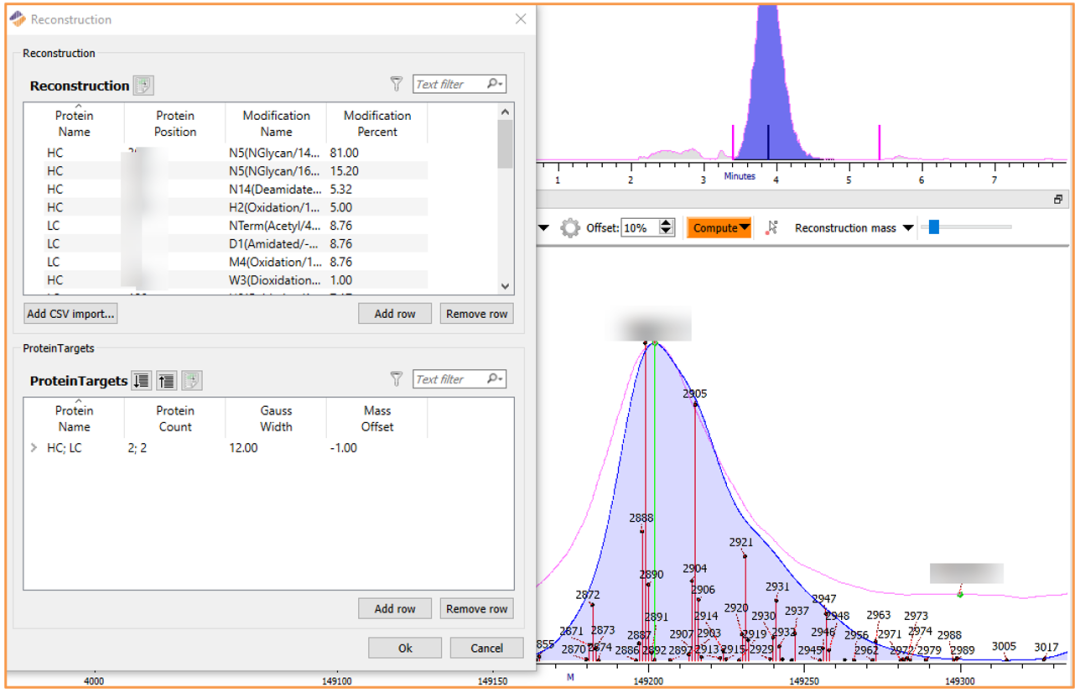
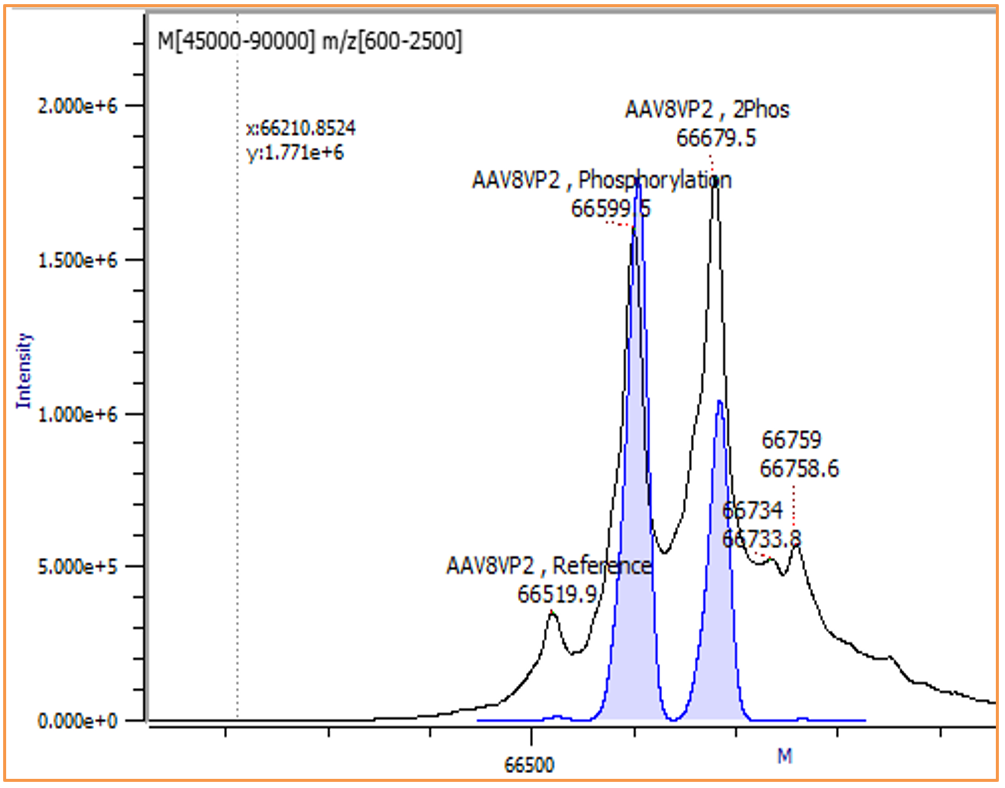
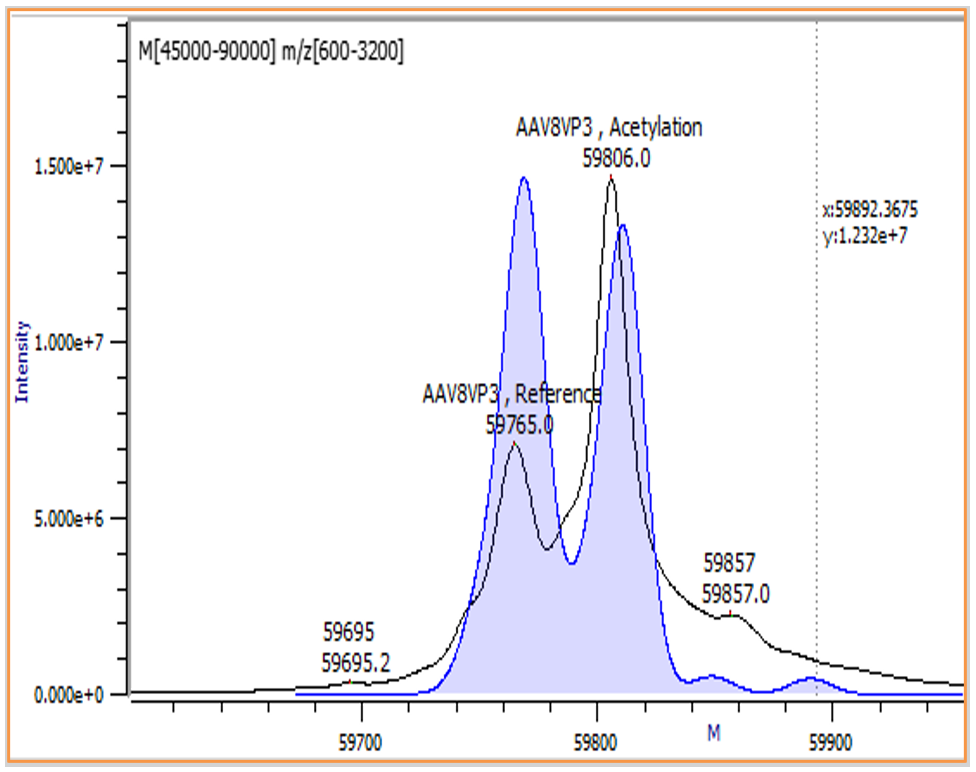
腺相关病毒(AAV)表征:
肽图法与完整分子量法的对比
Application Note
参考文献
Orthogonal Comparison of Analytical Methods by Theoretical Reconstruction from Bottom-up Assay Data.
Andrew C Nichols*, Yong Joo Kil, Andrew Mahan, Bo Zhai, Robert Hepler, Kristen Nields, Hirsh Nanda, Eric Carlson, and Marshall Bern.
Cite this: J. Am. Soc. Mass Spectrom. 2021, 32, 8, 2013–2018
Publication Date: March 25, 2021
https://doi.org/10.1021/jasms.0c00433
Copyright © 2021 American Society for Mass Spectrometry.
Published by American Chemical Society. All rights reserved.
关于Protein Metrics
Protein Metrics LLC是一家全球领先的质谱数据解析软件供应商,公司总部位于美国波士顿。我们为科研和企业用户提供高效准确的一站式质谱数据解析方案,帮助用户发现、解决问题。Protein Metrics在全球范围内提供销售和支持,目前已为超过200个企业和300个科研单位提供服务。
联系我们邀约演示:
王蕾 13482181958