Protein Metrics
高准编译
针对完整蛋白质分析和色谱工作流程的Trace Peak设置
简介
自定义洗脱峰设置,
可实现自动峰识别并简化工作流程,
适用于各种类型样品。
Intact, Oligo和Chromatogram模块均有此功能。

01
摘要

-
项目创建过程中的洗脱峰设置
-
导入Trace:嵌入式和非嵌入式Trace
-
选择Trace类型和Trace peak选项
-
自定义Trace peak选项
-
编辑Global Trace peak过滤器
-
创建项目后的洗脱峰设置
-
自定义编辑洗脱峰
-
手动编辑洗脱峰
-
编辑Global Trace peak过滤器
02
项目创建过程中的洗脱峰设置

导入Trace:嵌入式和非嵌入式文件
将原始文件拖放到 Samples Table 中后,任何嵌入式Trace将自动填充到Traces table(图 1)。
图 1.嵌入式Trace自动填充 Traces Table
如果 MS 控制软件未捕获到Trace,请将该Trace导出为 CSV 或TXT格式的 X, Y(强度, 时间)数据,添加到 Samples 选项卡上的对应行,如下所示(图 2)。
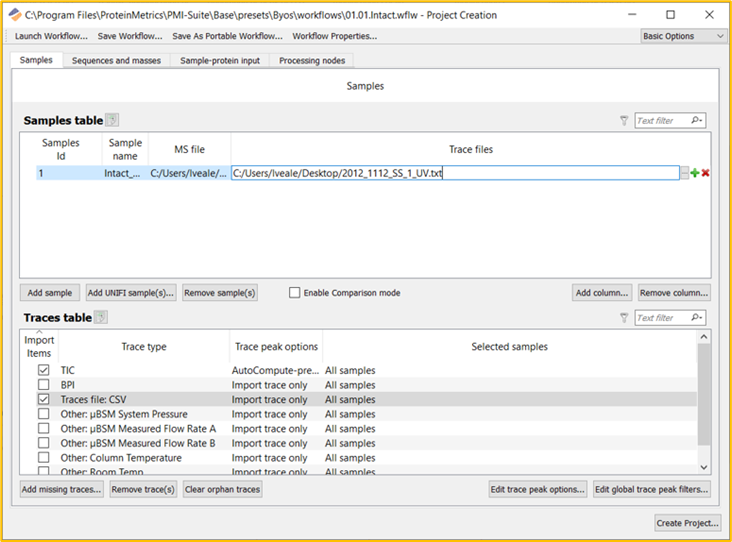
图 2.非嵌入式(导出)Trace填充 Traces Table
选择Trace类型和Trace peak选项
只需选中 Import Items 复选框,然后选择 Trace Peak Option(Trace peak选项),即可选择要去卷积或导入到项目中的Trace。提供多个可执行去卷积的Trace peak选项,包括 SingleSlice、AutoCompute、CustomSlice(s)、FixedWidthSlice(s) 或 AutoComputeByApex。最后一个选项 ImportOnly 允许导入Trace而不进行去卷积。更多信息详见下文。
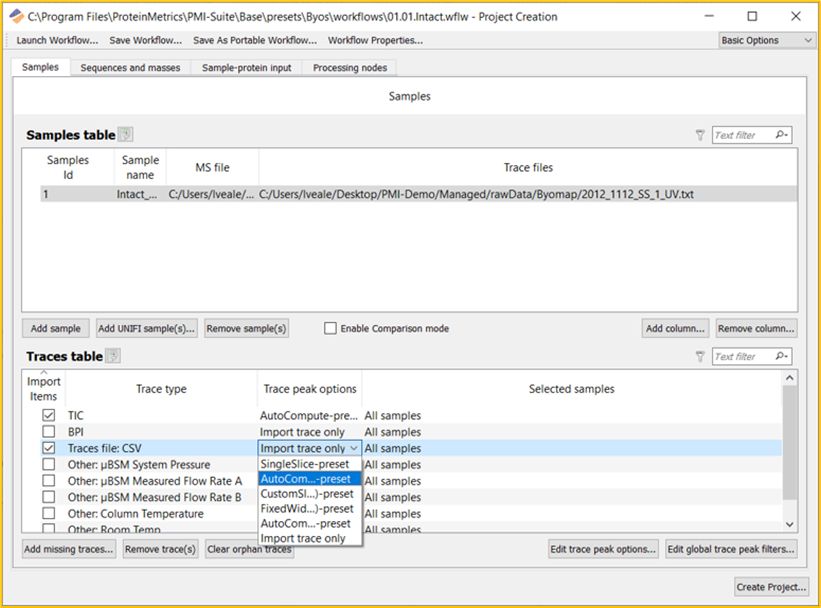
图 3.选择去卷积或导入到项目中的Trace
编辑Trace peak选项:自定义设置
可以在“Edit trace peak options…”按钮下找到自定义Trace peak选项。单击此按钮,将弹出菜单以供自定义。对于以下每项设置,可以将基线自定义为 Auto 或 Flat。在 Auto 基线下,可以设置平滑宽度。此处较大的平滑值可以合并Trace peak,而较小的值可能使峰分裂。
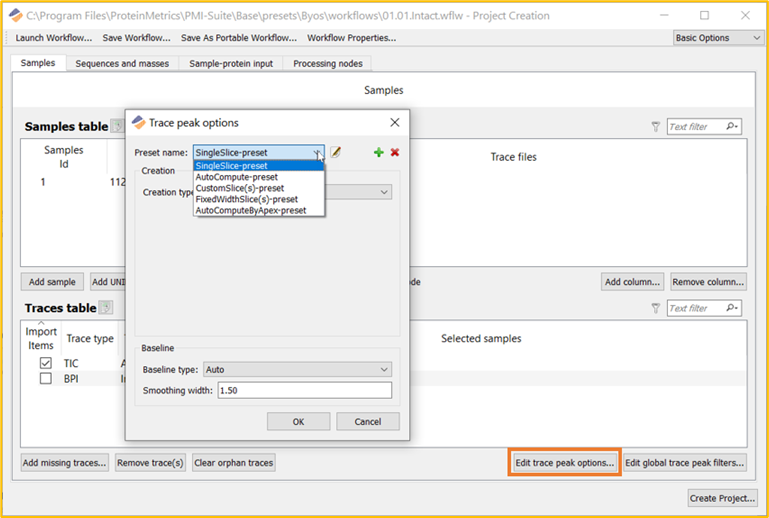
图 4.自定义Trace peak选项
自定义Trace peak选项:SingleSlice
SingleSlice Trace peak选项将整个样品处理为单个峰,针对每个文件显示一幅去卷积谱图。此设置对于简单的样品或“快速初步浏览”数据非常实用。
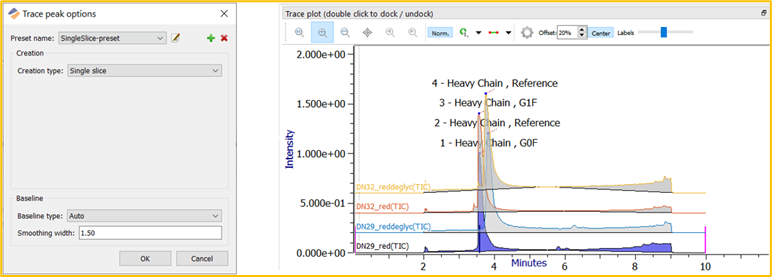
图 5.带Trace图示例的 SingleSlice 设置
自定义Trace peak选项:AutoCompute
AutoCompute Trace peak选项是默认的峰识别算法。该峰识别算法基于平滑后的最小宽度来确定Trace peak。此设置将针对每个峰显示一幅去卷积谱图。
为实现理想的峰识别,可能需要自定义平滑宽度和最小宽度。在以下示例中(图 6),将平滑宽度设置为每 0.1 分钟查找一个拐点。最小宽度设置为 0.5 分钟。 对于宽的起伏峰,应增大这些设置。同样,对于窄而尖的峰,可能需要减小这些设置。
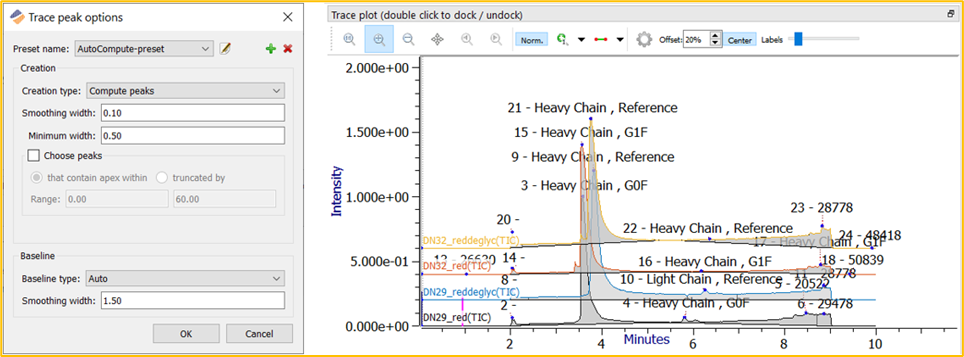
图 6.带Trace图示例的 AutoCompute 设置
自定义Trace peak选项:CustomSlice(s)
CustomSlice(s) Trace peak选项使分析人员能够定义开始和结束时间点,针对每个时间点显示一幅去卷积谱图。此设置非常适用于 IdeS 酶解物或预期峰在特定时间窗口内洗脱的样品。
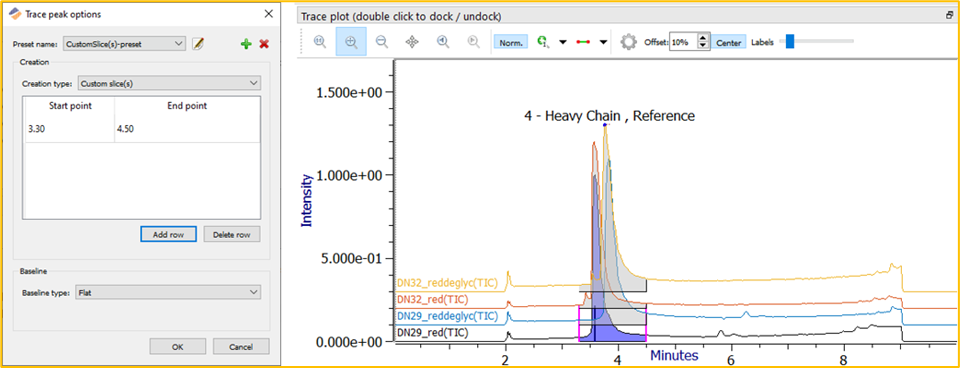
图 7.带Trace图示例的 CustomSlice(s) 设置
自定义Trace peak选项: FixedWidthSlice(s)
FixedWidthSlice(s) Trace peak选项将Trace peak按设定的间隔分开。此设置将针对每个间隔显示一幅去卷积谱图。FixedWidthSlice(s) 非常适用于色谱分离结果不佳的样品(例如 AAV)。
在下面的示例中(图 7),将第一个片段(间隔)设置为 3.25 分钟至 3.50 分钟。将片段之间的步长设置为 0.25 分钟,或与第一个片段相同的持续时间以免片段重叠。请注意,减小片段之间的步长以实现一定程度的重叠,可能有利于特别复杂的样品。最后,将 Number of slices 设置为 10。这相当于 10 个片段,每个 0.25 分钟,或针对 2.5 分钟的片段。
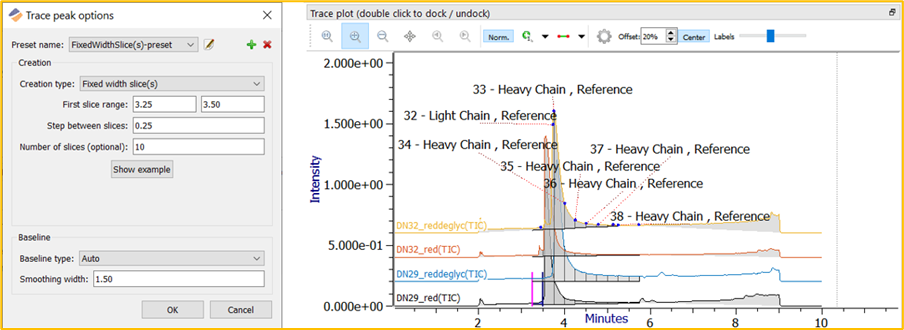
图 8.带Trace图示例的 FixedWidthSlice(s) 设置
自定义Trace peak选项:AutoComputeByApex
AutoComputeByApex Trace peak选项的运行方式与默认 (AutoCompute) 的峰识别算法相同。有关峰识别自定义详细信息,请参见上文。
此设置可以更精细地控制色谱时间范围。这两个选项是“that contains apex within”或“truncated by”。
如果峰肩部或尾部落在指定的时间范围之前或之后,“that contains apex within”将选中整个峰(图 9a)。
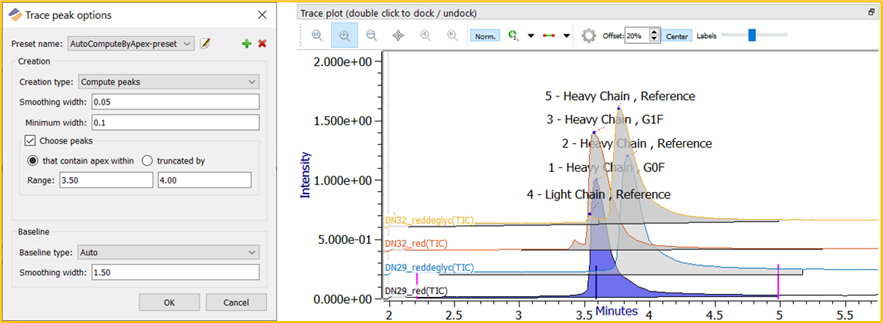
图 9a.带Trace图示例的 AutoComputeByApex 设置 — that contains apex within
或者,“truncated by”设置将执行硬截止,并排除指定时间范围之前或之后的任何峰肩部或尾部(图 9b)。
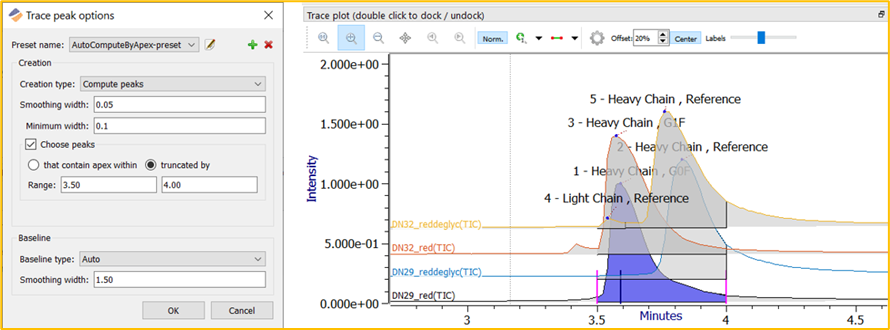
图 9b.带Trace图示例的 AutoComputeByApex 设置 — truncated by
编辑Global Trace peak过滤器
屏幕右下角的“Edit global trace peak filters”按钮可以更全面地控制Trace peak。
峰选项部分允许进行可选的时间截止。此外,可应用最小峰面积阈值来跳过低丰度峰。默认情况下,此阈值设置为 5%。如果发现丢失低丰度峰或片段,则可以将该过滤器调整为更低的百分比,或将其设置为零。
峰比较选项用于批次间样品对比。默认情况下,任何高于或低于参比峰 10 %的Trace peak都将以红色突出显示。
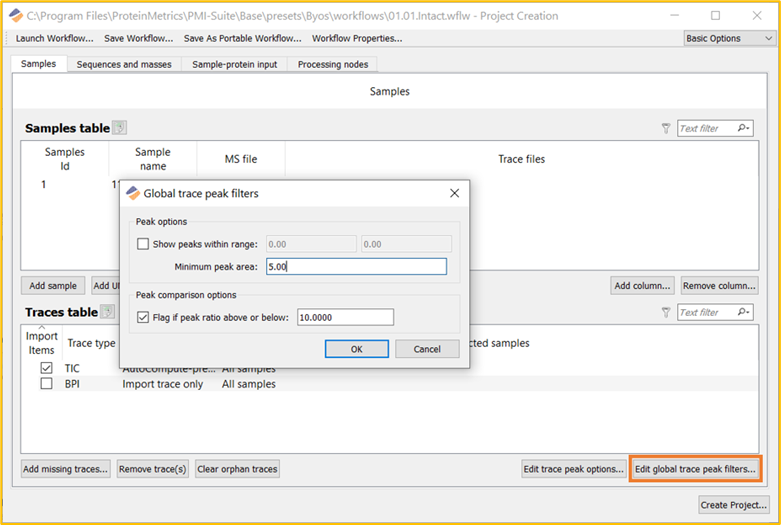
图 10.GlobalTrace peak过滤器
03
创建项目后的洗脱峰设置调整

自定义编辑洗脱峰
在创建项目后调整所有样品的洗脱峰的最快速、最简单的方法是在 Edit> Recompute trace peaks and baseline… 下进行操作(图 11)。将打开自定义Trace peak的弹出窗口以供调整。Global编辑Trace peak后,转至 Edit> Auto compute,并分配质量数以对Trace进行Global去卷积。
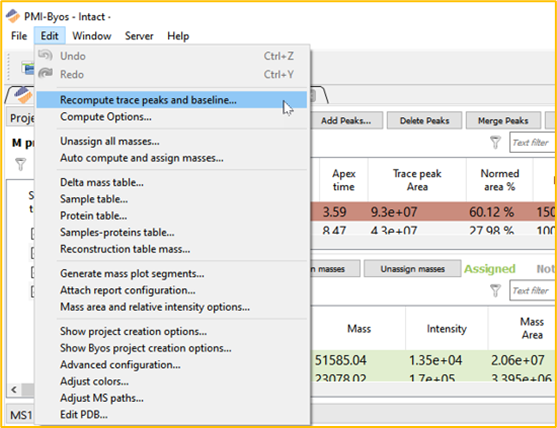
图 11.在创建项目后访问 Recompute trace peaks and baseline 菜单
手动编辑洗脱峰
在一些情况下,有必要手动调整Trace peak。在Trace peak表上方,提供 Add peaks、Delete peaks、Merge peaks 和 Split peaks 按钮。通过单击并编辑Trace peak表中的时间,或拖动Trace图色谱图上的粉色条,可以编辑峰的开始和结束时间。
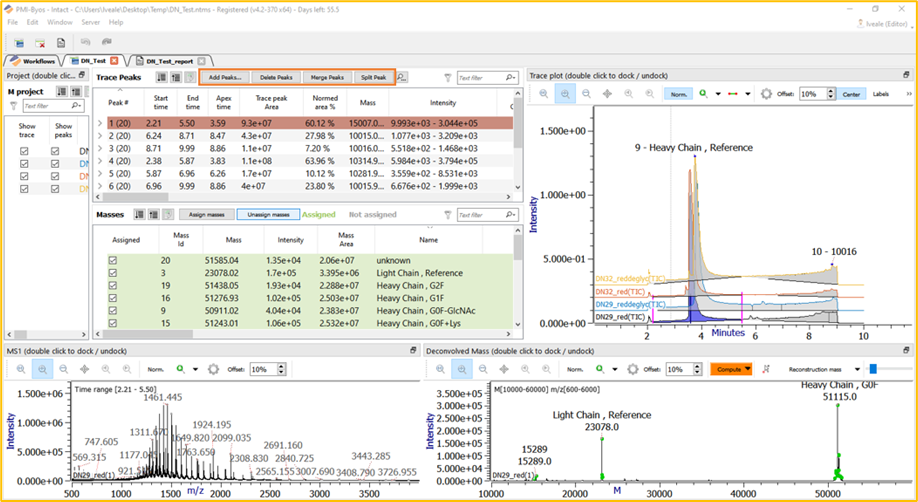
图 12.手动编辑洗脱峰
编辑GlobalTrace peak过滤器
创建项目后,单击Trace peak表上方的放大镜按钮即可访问Global Trace peak过滤器菜单。在这里,可以编辑可选时间范围、最小峰面积阈值和比值标记。
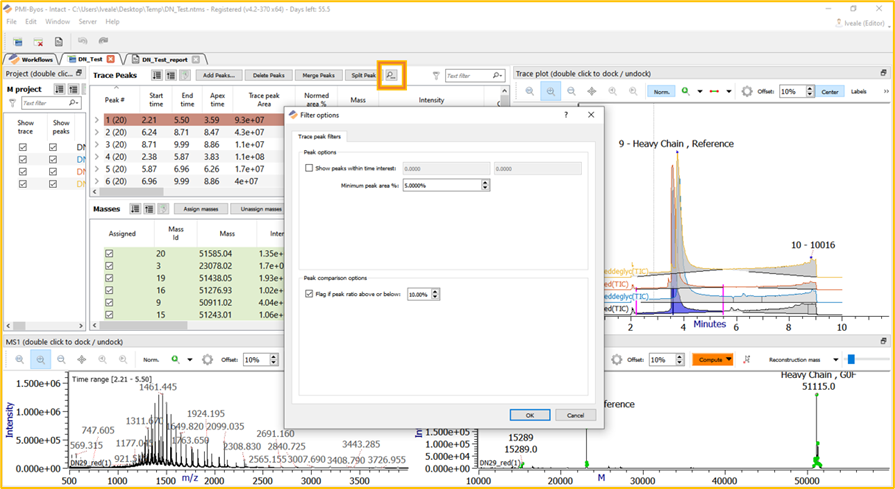
图 13.编辑GlobalTrace peak过滤器
有关完整蛋白质分析和色谱数据分析工作流程的更多资源和背景信息,请访问 https://www.proteinmetrics.com/workflows/
或通过 support@proteinmetrics.com 联系客户成功团队。
关于Protein Metrics
Protein Metrics LLC是一家全球领先的质谱数据解析软件供应商,公司总部位于美国加州。我们为科研和企业用户提供高效准确的一站式质谱数据解析方案,帮助用户发现、解决问题。Protein Metrics在全球范围内提供销售和支持,目前已为超过150个企业和300个科研单位提供服务。
联系我们邀约演示:
王蕾 13482181958


