摘要

目的:使用靶向连接子酶切和自动化数据分析工具对融合蛋白进行Middle-down分析。新一代生物制药结构通常包含具有多个 GS 连接子的结构域以抵抗蛋白水解。使用 GlySERIAS™ 蛋白酶酶切融合蛋白 Blinatumomab 的富甘氨酸连接子,并通过 LC-MSMS 分析所得的片段。采用 Byos® 的Intact Workflow自动去卷积并鉴定碎片质量数。利用Middle-down测序来确认酶切位点。
简介和策略
融合蛋白可结合多个蛋白结构域,从而解决特定治疗难题。通常,这些结构域使用包含多个甘氨酸残基(G)的连接子或穿插有丝氨酸 (S) 的(GS) 连接子。这些连接子专为抵抗蛋白酶降解而设计,因此难以通过传统的 LC-MS 方法进行分析。
Middle-down策略:
GlySERIAS™ 用于水解融合蛋白 Blinatumomab 中的柔性连接子。使用固定化酶,使连接子酶切更完全。
使用 Bruker Impact II QTOF 采集LC-MS/MS数据 ,在 Protein Metrics Intact Workflow中处理原始数据文件。
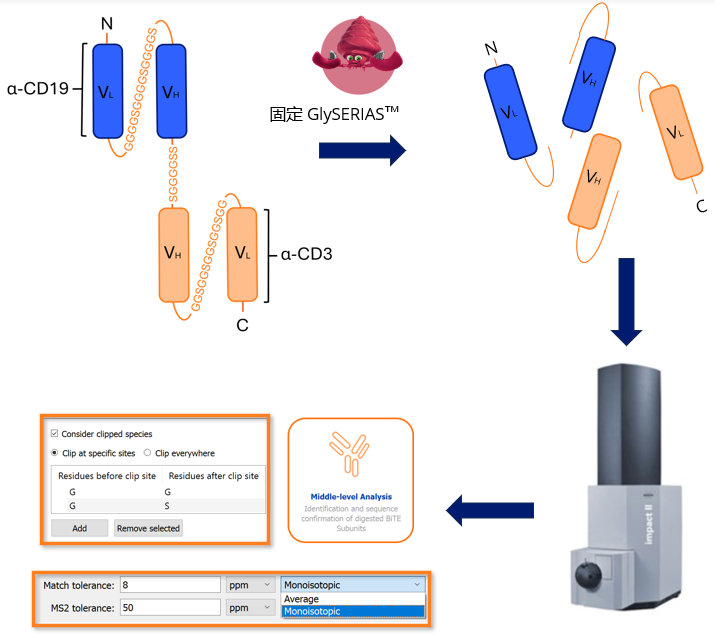
酶切片段的鉴定和序列确认
图 1.Byos Intact Workflow(UI)。该工作流程能够高效鉴定和验证通过 LC-MS 分离的主要蛋白水解片段。利用Clip功能成功鉴定了 VL 和 VH 亚基的Clip形式。通过 MS2 谱图 (D) 的注释确认了酶切位点。
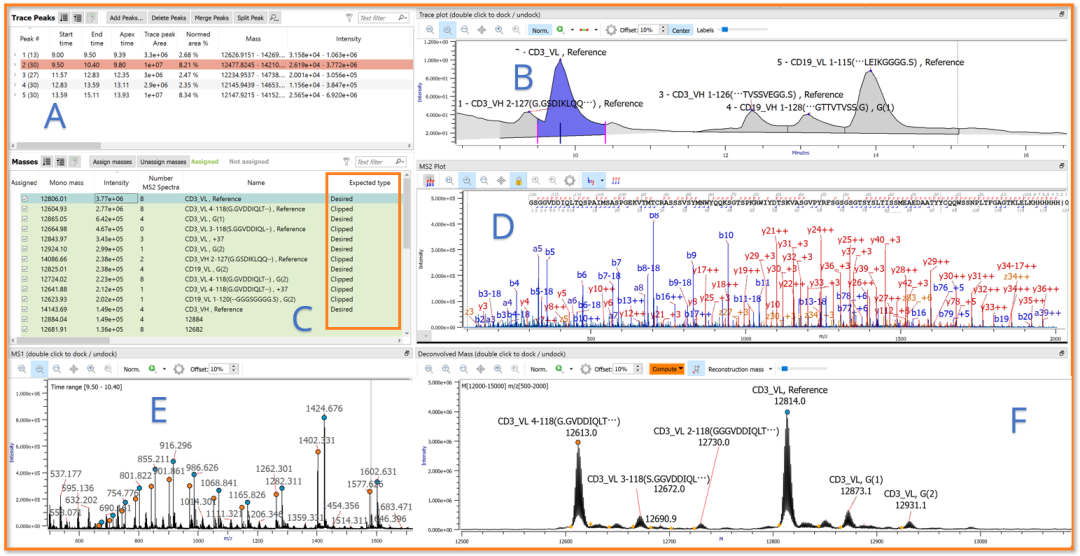
A. Trace Peaks – 汇总了Trace plot中4个积分峰。
B. Trace plot – 显示可手动编辑峰边界的 TIC。
C. Masses – 峰 1 中发现的去卷积质量数。
D. MS2 Plot – 亚基 CD3_VL 的注释 MS2 谱图,确认序列和酶切的位置。
E. MS1 – 图中彩色小圆点将多重电荷态的m/z与Deconvolved Mass中的中性质量峰相关联。
F. Deconvolved Mass – 去卷积时仍保持同位素分辨率。用黄色菱形标记的单同位素质量数。
鉴定概要
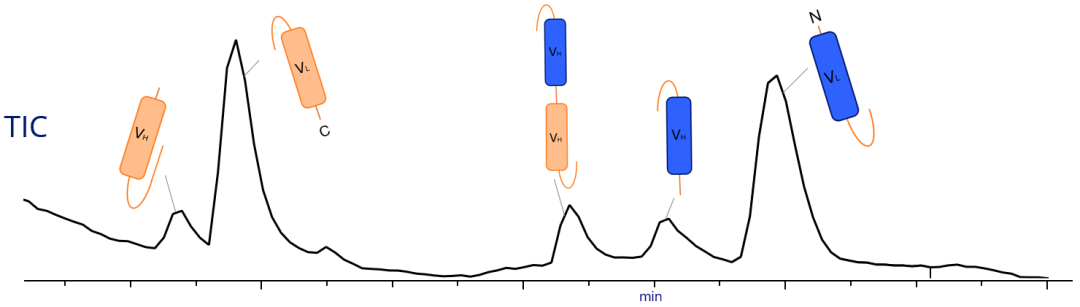
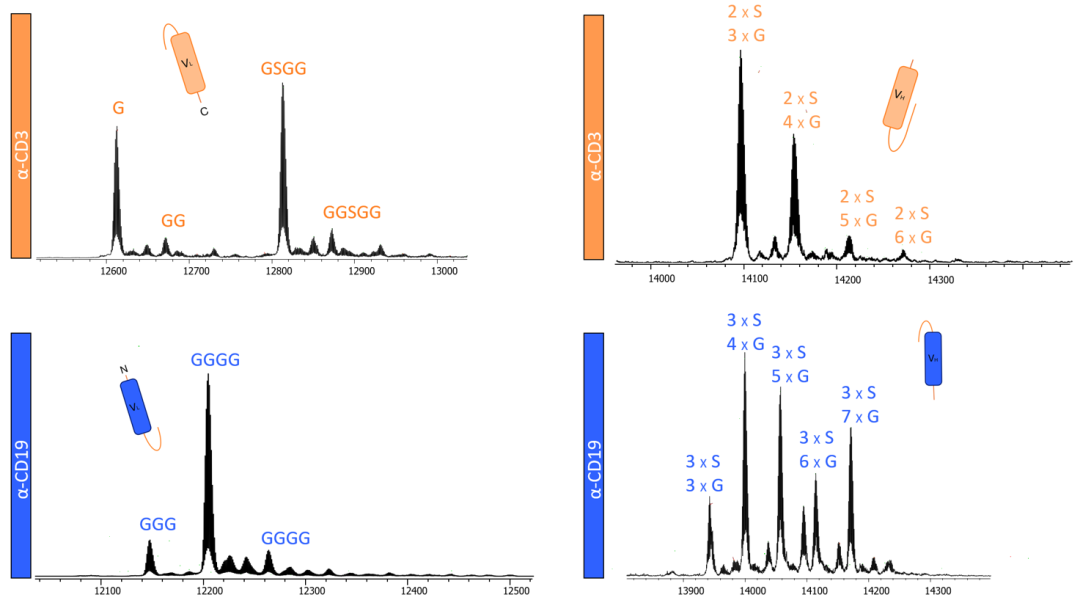
通过 GlySERIAS 酶切得到的亚基异质性混合物主要通过去卷积 MS1 数据的单同位素质量数分配进行鉴定。利用 Byos Intact Workflow中的Clip功能(可自动检测 GG、GS 和 SG 残基之间的酶切),自动搜索所有可能的Clip片段。
Byos Intact Workflow支持Middle-down分析,可通过注释 MS2 数据确认所有主要亚基的序列。这反过来提供了更可靠的酶切位点分配。
与Intact Workflow的Top-down分析相比,Middle-down策略与 GlySERIAS 联用后,蛋白质片段更易于通过 MS2 进行分析。


利益冲突证明
部分作者是 Genovis 的员工和/或股东,Genovis 已经将本文所述的酶商业化。
关于Protein Metrics
Protein Metrics LLC是一家全球领先的质谱数据解析软件供应商,公司总部位于美国加州。我们为科研和企业用户提供高效准确的一站式质谱数据解析方案,帮助用户发现、解决问题。Protein Metrics在全球范围内提供销售和支持,目前已为超过150个企业和300个科研单位提供服务。
联系我们邀约演示:
王蕾 13482181958