Byos软件内置大量的聚糖信息,如聚糖库,聚糖组成,聚糖名称,聚糖卡通图,聚糖分组等等,允许用户自定义的添加和修改。
修改命名法
Byos软件中默认的聚糖命名法是生物制药命名法,如G1F, G2F等。如果想切换成牛津命名法,如FA2G1, FA2G2等,具体方法如下:
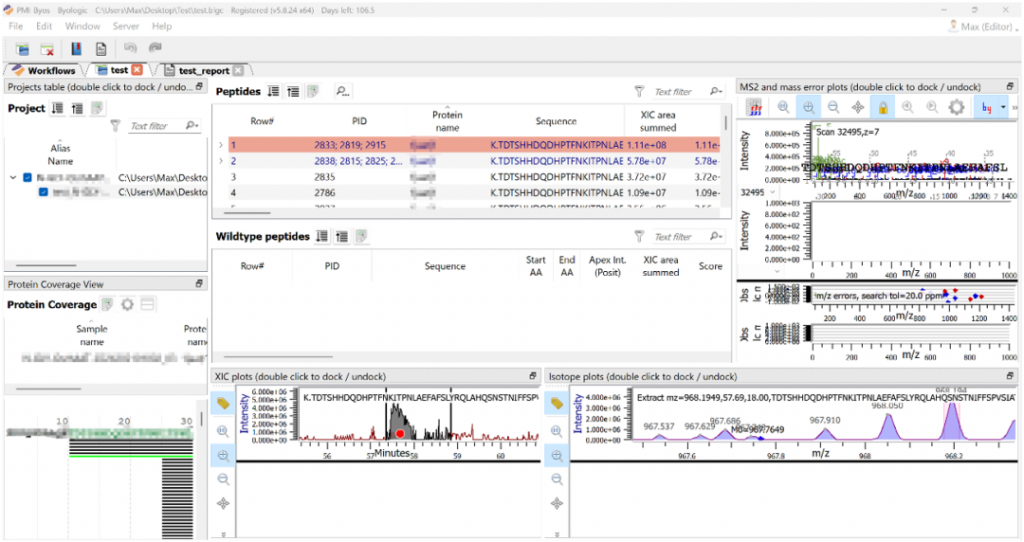
第一步:正常创建项目,选择合适的参数进行设置,点击Create Project,完成数据的分析步骤,得到下图可视化界面及报告界面。
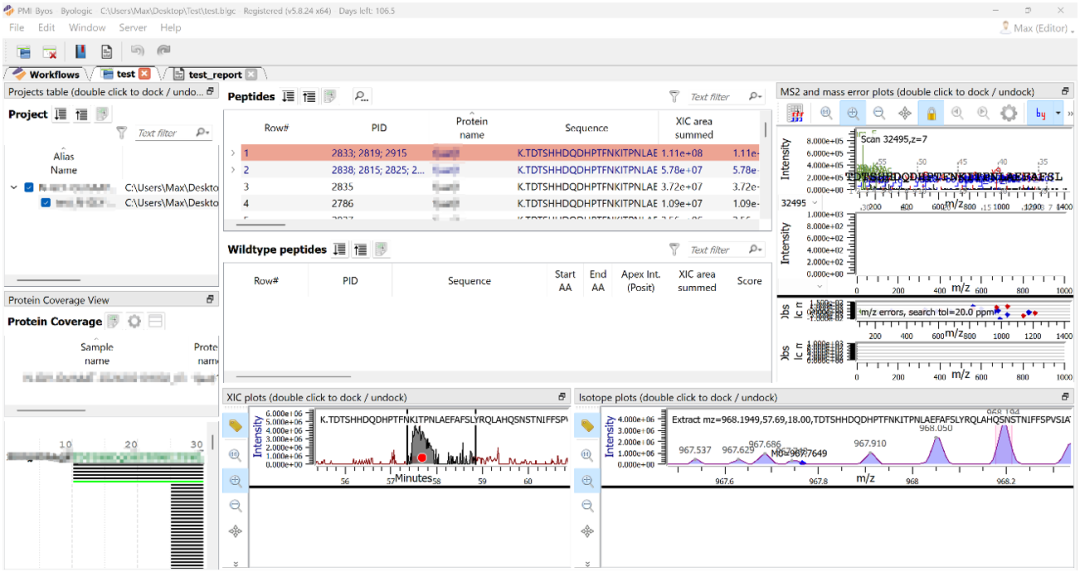
图1
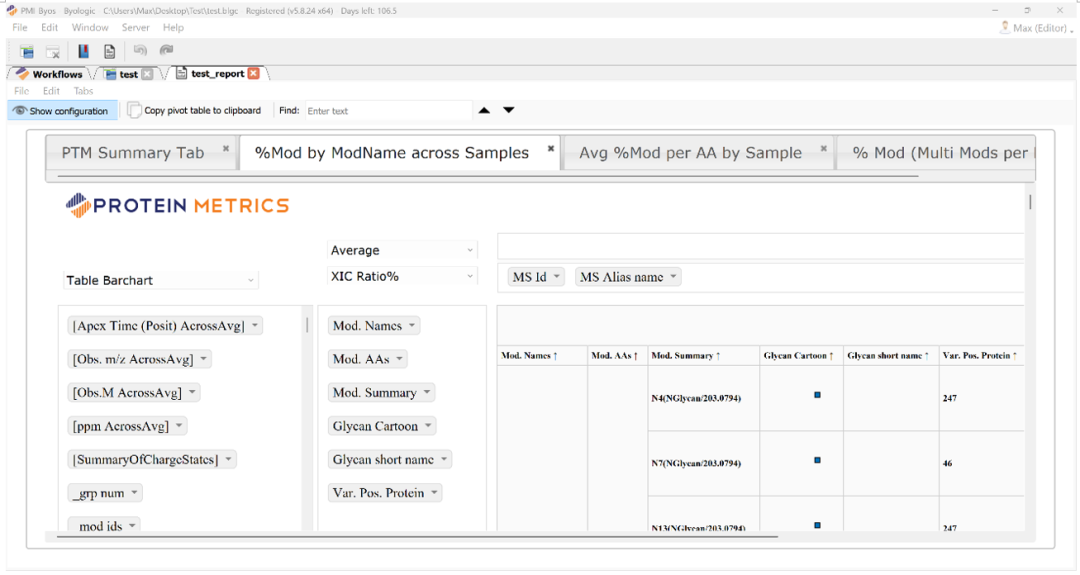
图2
第二步:在图1的分析界面中,首先进行数据筛选,选择需要在报告中展示的信息,例如只关心糖肽,通过下图🔍选项,进行Glycosylation的筛选。
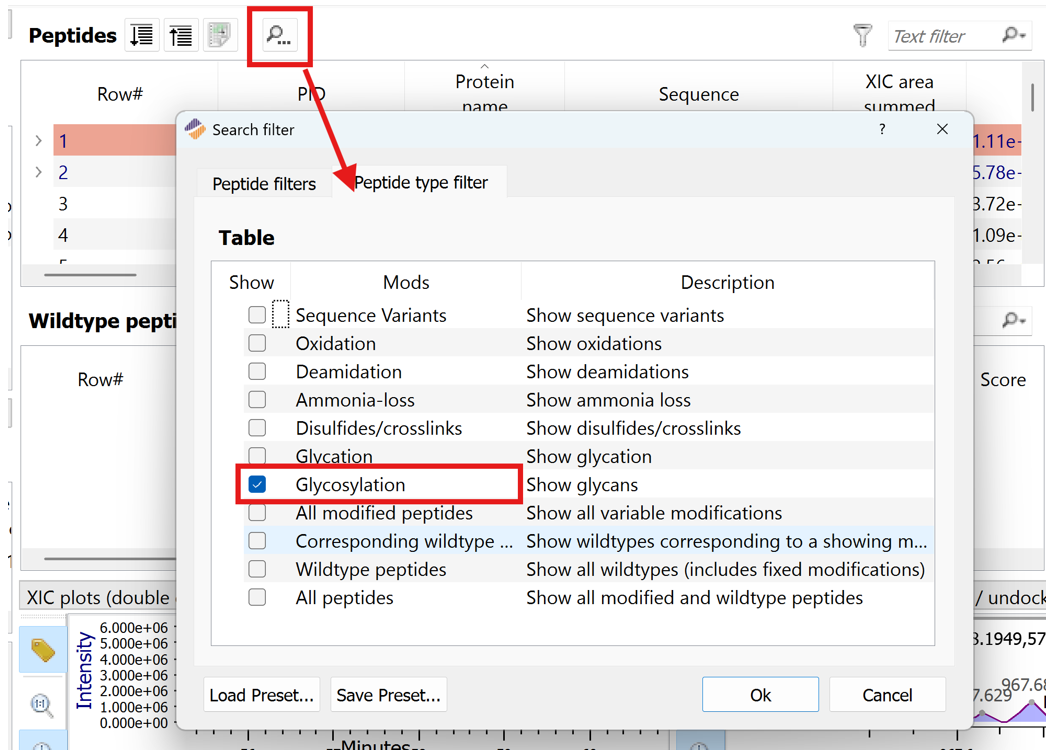
图3
第三步:调取Pre pivot column。
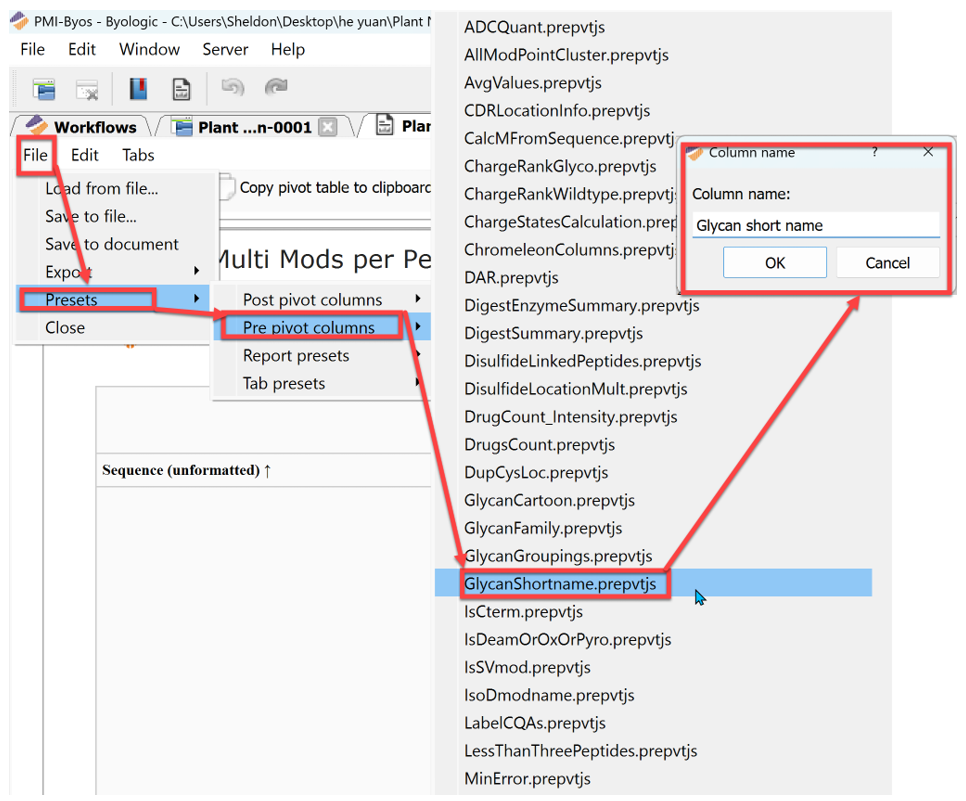
图4
第四步:将调取出的Glycan Short Name的表头columns拖拽至展示columns中。
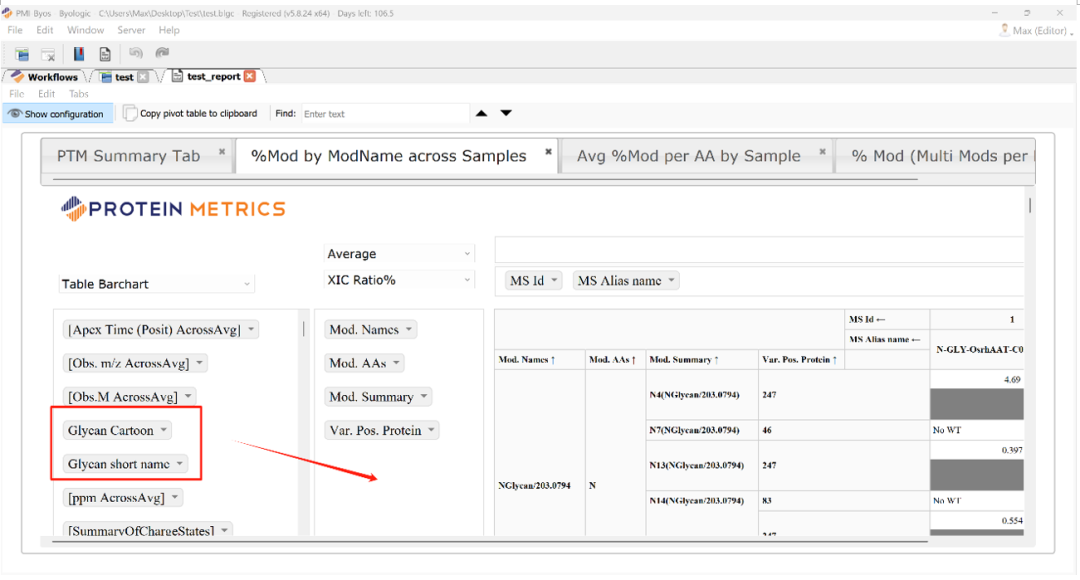
图5
第五步:报告中自动添加Glycan short name信息。
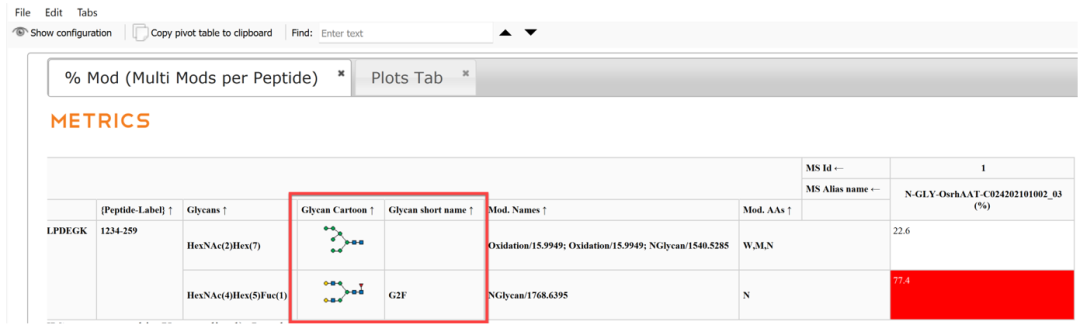
图6
第六步:修改命名法和添加未收录的糖型。
A:打开Glycan short name脚本,点击✏处
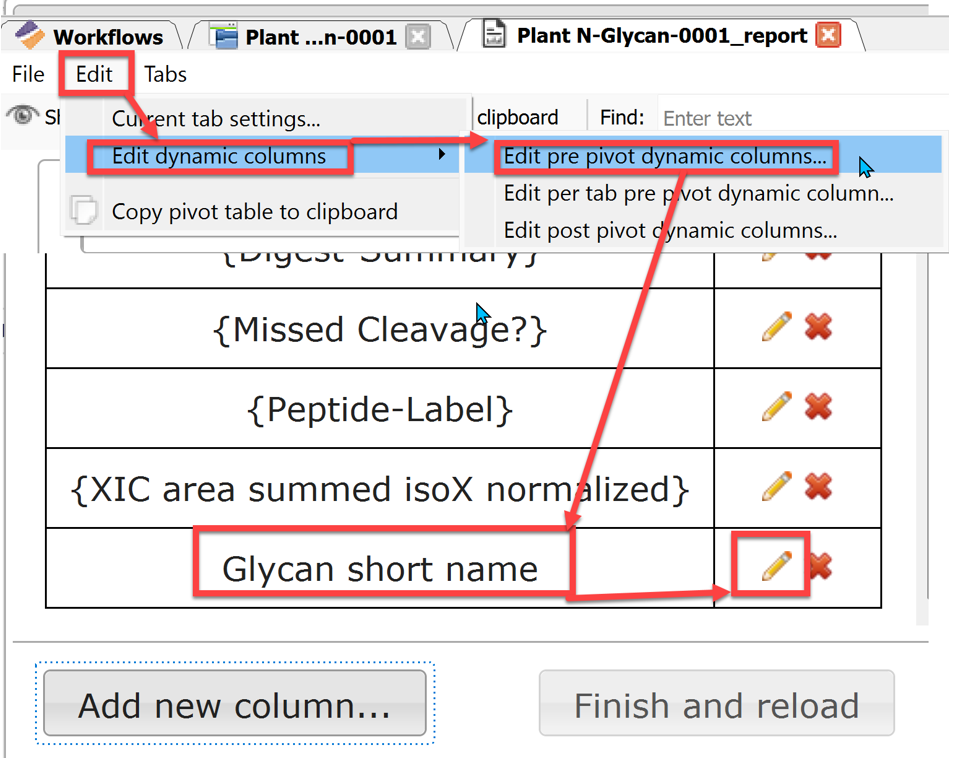
图7
B:将红色方框内生物制药名称替换成牛津名称
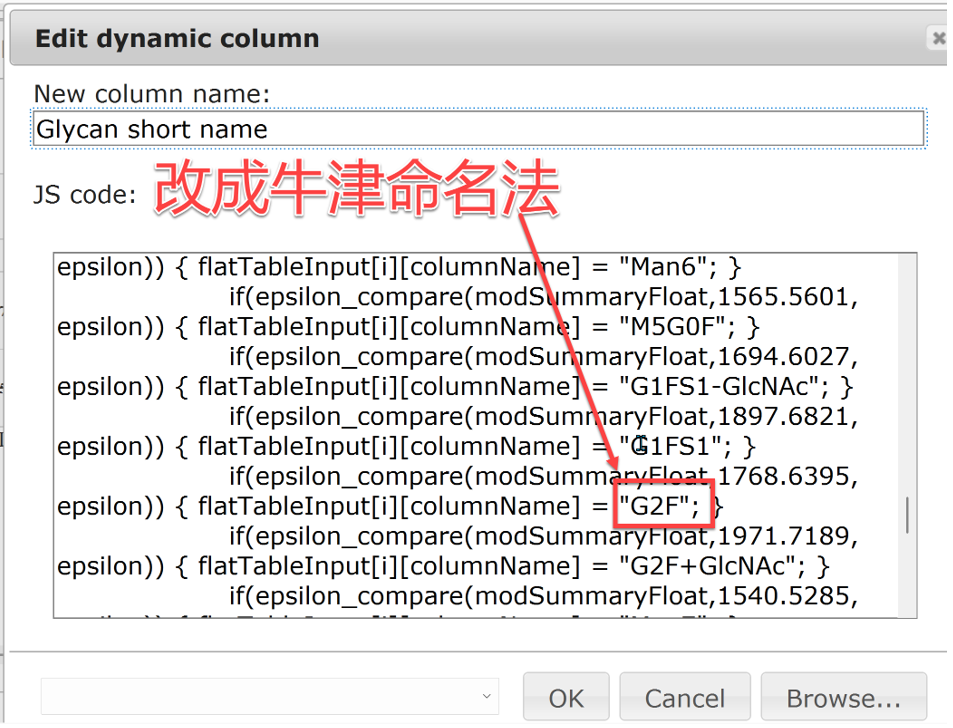
图8
C:添加未收录糖
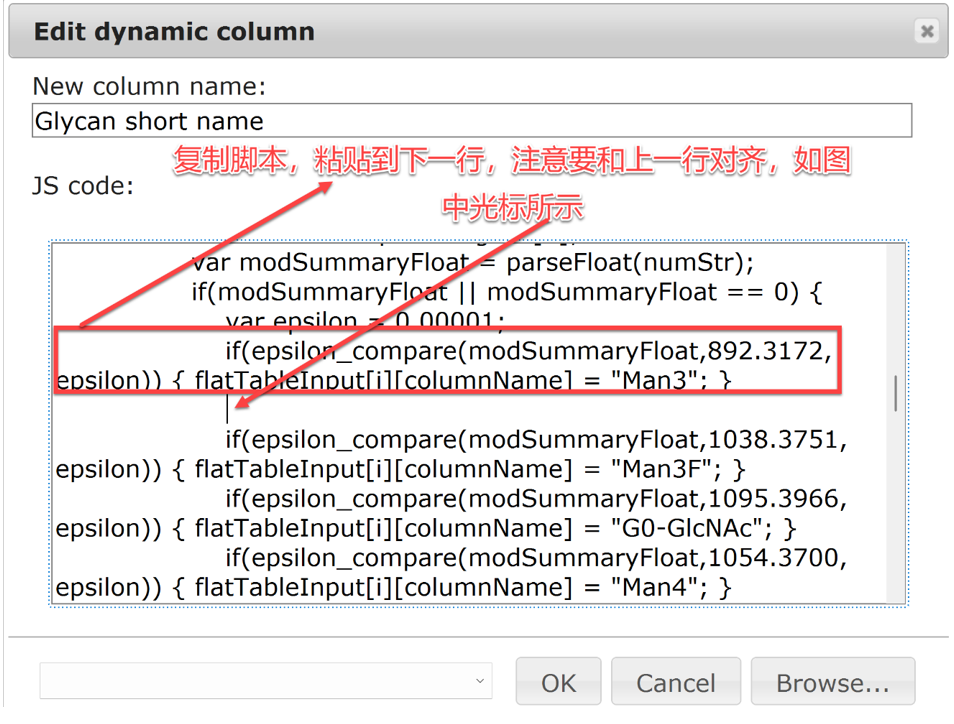
图9
D:更新report,可看到更新后的聚糖名称
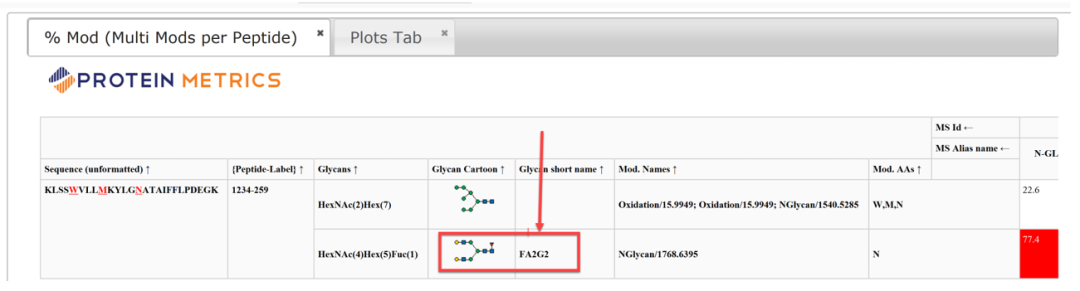
图10
创建卡通图
Byos中的聚糖卡通图是按照IUPAC命名法绘制而成,支持自定义添加卡通图。
第一步:查看卡通图。
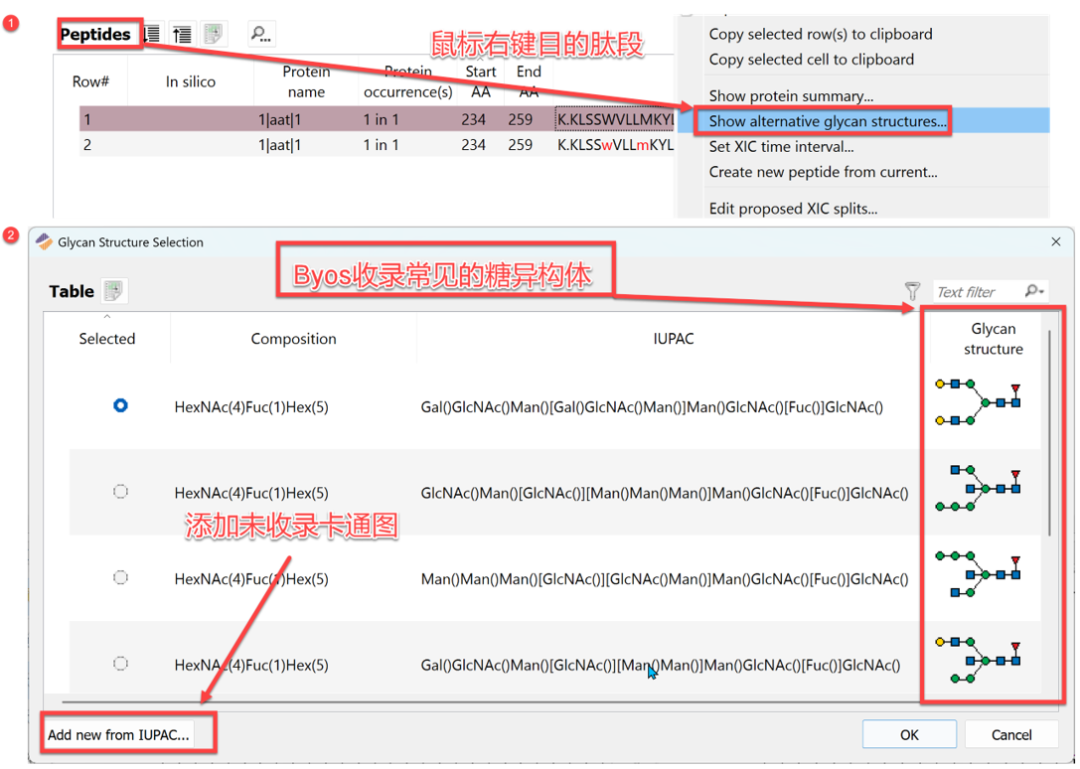
图11
第二步:如果没有看到卡通图,说明软件没有收录,需要手动添加。
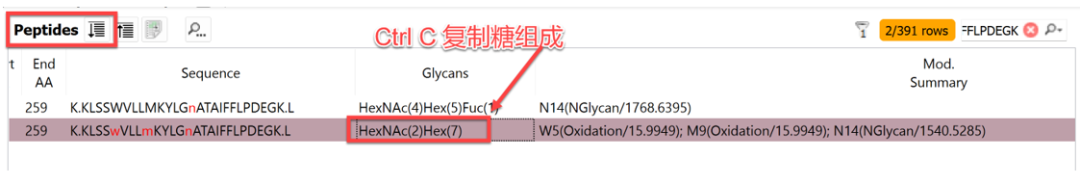
图12
第三步:登陆网站 “Structures”
网址: https://glyconnect.expasy.org/browser/structures
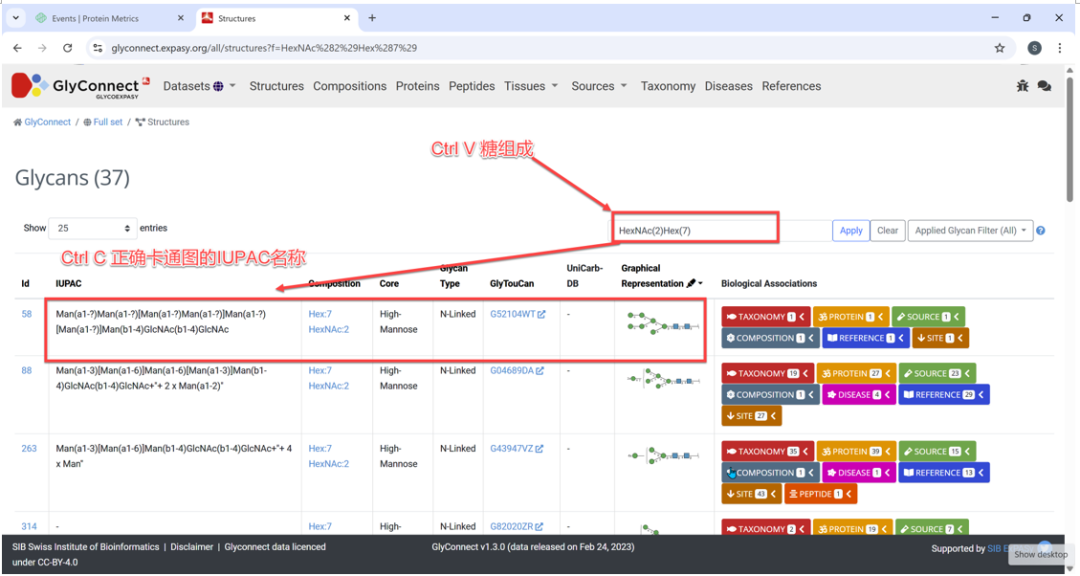
图13
第四步:添加新卡通图。
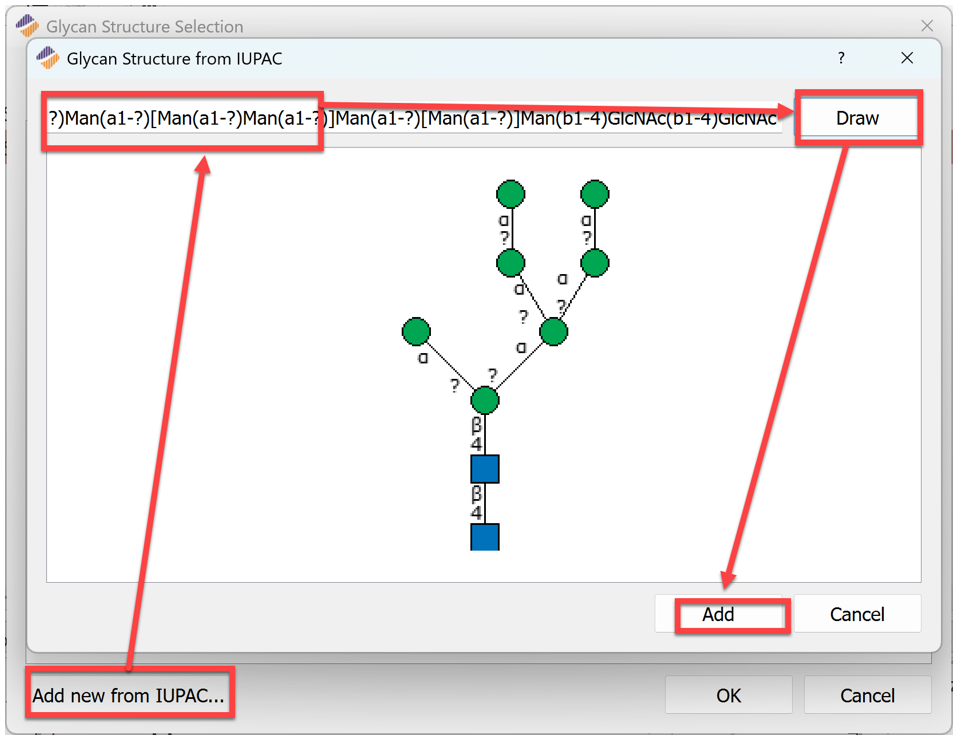
图14
第五步:IUPAC命名法中含有单糖的连接方式,如上图所示β 4(表示β 1,4糖苷键,1往往会省略),α ? (表示α 1,?糖苷键,?表示未知位点)等。如果不想显示连接信息,可以删除IUPAC名称中括号内的字符。
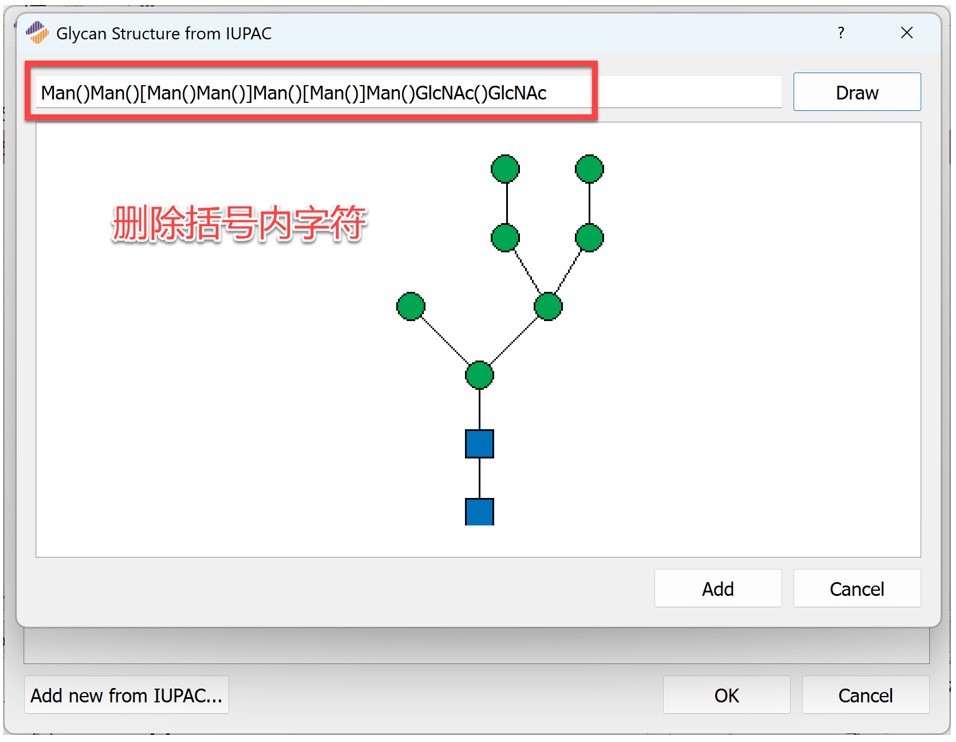
图15
第六步:选中新添加的糖卡通图,更新report。
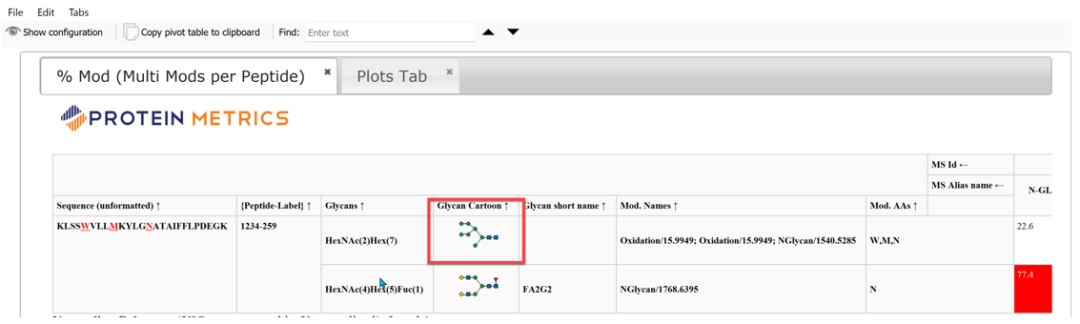
图16
小结
Byos软件收录大量的pre和post pivot脚本,允许用户自定义添加和修改,从而设计专属的个性化报告模板。
关于Protein Metrics
Protein Metrics LLC是一家全球领先的质谱数据解析软件供应商,公司总部位于美国波士顿。我们为科研和企业用户提供高效准确的一站式质谱数据解析方案,帮助用户发现、解决问题。Protein Metrics在全球范围内提供销售和支持,目前已为超过200个企业和300个科研单位提供服务。
联系我们邀约演示:
王蕾 13482181958