引言
mRNA药物质量属性 (PQAs) 的表征分析对于确保其安全性和有效性至关重要。利用LC-MS/MS技术进行酶解寡核苷酸序列映射,能够在单一实验中同时表征5’端帽子结构、寡核苷酸序列以及3’端多聚腺苷酸尾巴。
方法
eGFP的mRNA经hRNase4酶解后,采用IP-RPLC-MS技术进行分析。
实验使用Phenomenex bioZen2.6 µm Oligo 150 x 2.1 mm色谱柱 (PN:00F-4790-AN),采用70分钟的离子对反相液相色谱梯度洗脱程序,并在Thermo Q-Exactive Plus质谱仪上以DDA负离子模式进行检测。
数据处理则使用Byos Digested Oligonucleotide和Oligo软件(V5.6)完成。
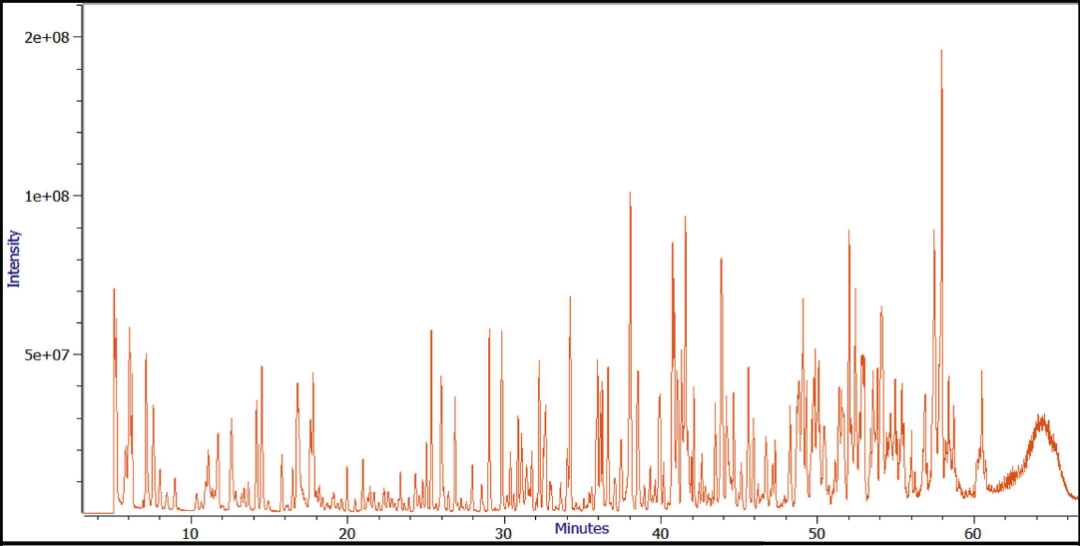
图1. hRNAse4酶解eGFP的总离子流色谱图(TIC)

图2. eGFP和mRNA的序列覆盖图
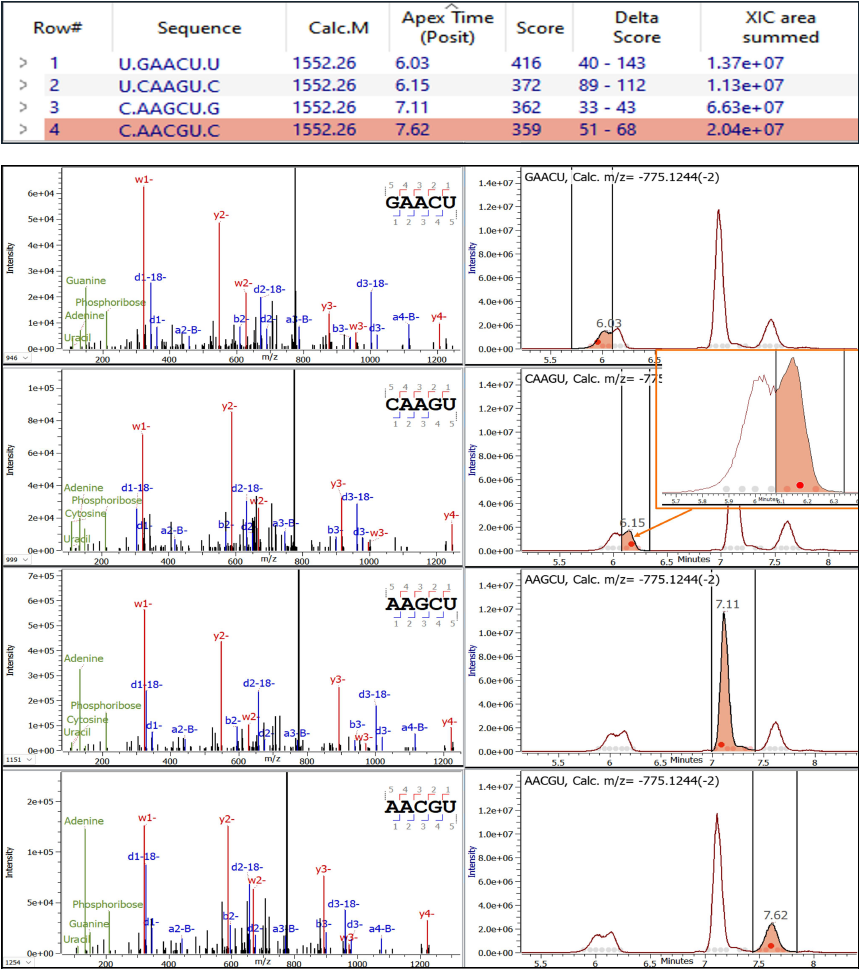
图3. 四种寡核苷酸异构体的二级质谱图及提取离子流色谱图。上方的寡核苷酸表格包含了有助于评估寡核苷酸鉴定结果的列。图表和表格均为Byos Digested Oligonucleotide分析视图的一部分。
结论
寡核苷酸序列比对,5’cap及3’PolyA Tail
>99%的eGFP序列覆盖率(见图2)。
Byos软件通过二级质谱 (MS/MS) 自动分配寡核苷酸异构体 (见图3)。IPRP分离对于可靠地分配异构体至关重要。
XIC中的灰色/红色圆点表示异构体的寡核苷酸序列标记物。灰色圆点代表已分配的OSM异构体。(见图3左侧放大图)。
Delta Score: 已分配寡核苷酸与次优得分寡核苷酸/异构体(即异构体的“独特性”得分)之间的二级质谱 (MSMS) 得分差异。
在注释的二级质谱图中观察到的用于区分关键异构体的碎片离子。
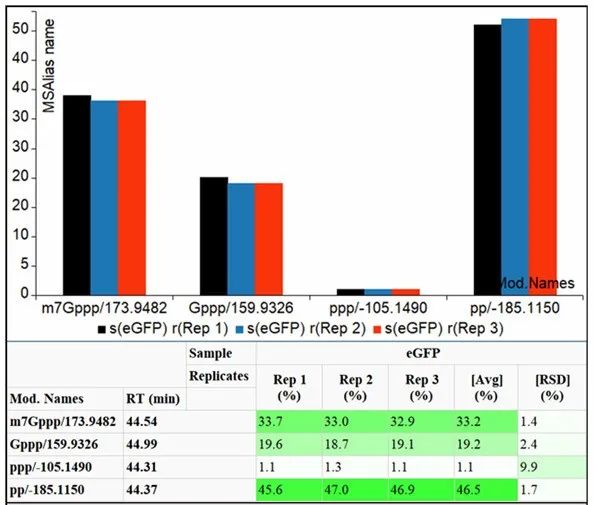
图4. 使用Byos报告功能生成的条形图和表格,展示了5’cap各类型的相对丰度(n=3,即样本量为3)
m7Gppp 5’cap结构的相对丰度为33.2%,%RSD=1.4% (n=3)。(见图4)
观察到的5’cap结构中间体包括Gppp (占比19%)、ppp (占比1%) 以及pp (占比47%)。(见图4)
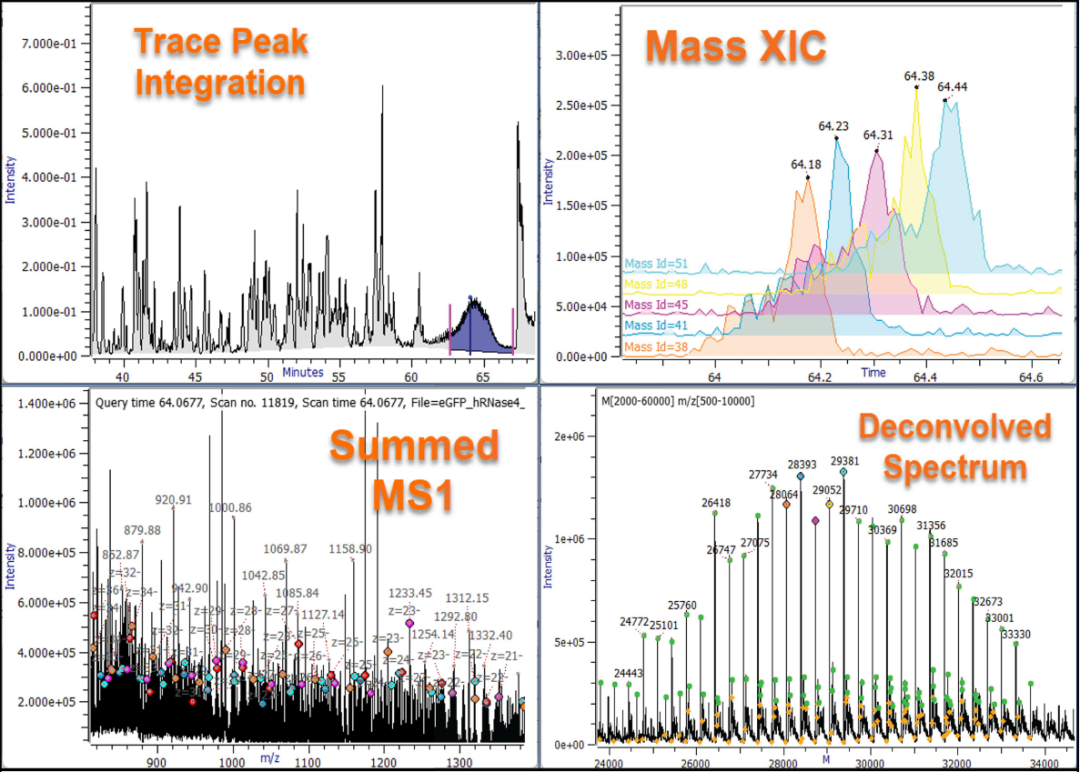
图5. Byos Oligo软件对eGFP多聚腺苷酸尾巴进行的一级质谱扫描数据求和、解卷积处理以及Mass XIC的绘制。
在解卷积后的质谱图中,多聚腺苷酸尾巴长度为51至84的序列实现了质量匹配。(见图5)
Mass XIC图展示了多聚腺苷酸尾巴在LC中的分离效果。(见图5)
作者
Steven Broome1, Roxana Eggleston-Rangel2, Maria Basanta-Sanchez1, Marshall Bern1
1 Protein Metrics, Boston, MA
2Phenomenex, Torrance, CA
END

