
使用 Byos 软件的自动标记功能
提高数据审查效率
特此感谢:
– Protein Metrics Research & Development and Customer Success Teams, Cupertino, CA, U.S.A
摘要
• 本文集阐述了:如何使用 Byos 软件来提高生物治疗药物表征工作中,LC-MS/MS分析数据的审查效率。
• 对于 Byos 中的序列变异体和翻译后修饰分析,我们在此展示了自动注释生成功能,进而表明了分配数据的置信水平。
• 借助这些注释,审查者可以快速列出真实的阳性结果,排除错误的数据,并将其工作重点放在需要审查的数据上。
• 此外,这些注释可以提高数据审查的一致性,因为每个项目都以相同的方式进行处理,使用自动标记来处理类似问题。
简介
近年来,数据分析在自动化方面取得了巨大进步,但有时仍然需要手动管理数据。数据审查通常比较枯燥乏味且耗时甚巨,并且可能会根据针对样本的解释引入诸多变数。为了节省时间并消除不同分析人员造成的差异,应迅速排除基于已知误读造成的错误识别。
在本文集中,我们展示了 Byos 如何根据专有的 2 个既定workflow-序列变异体分析和翻译后修饰分析而设计的决策树自动标记结果。结果标记中有任何关键问题,任何未显示问题的数据均标记为问题已清除。我们将这些标签称为“验证器”,它们可用于提高数据分析的效率。
生物治疗药物生产过程中发生的意外氨基酸置换、插入和缺失(序列变异体)可能会导致最终产品中出现杂质。序列变异体的检测和表征非常具有挑战性,因为他们通常在产品中的表达水平较低。这可能需要花费数小时的时间进行手动数据检查,从而对检测结果进行验证。使用 Byos 序列变异体验证器,可以简化排除误报和验证真实标识的过程。
此外,生物治疗产品翻译后修饰的表征对于了解其功效和稳定性至关重要。对于降解研究,例如本文集中示例的分析样品,经常要对不同应力条件下的修饰进行比较,以评估产品稳定性。在此过程中,通常会检测到氧化和脱酰胺。翻译后修饰的数据审查也可以使用 Byos 修饰验证器进行数据简化。
实验条件
对NIST Mab 的对照样品和应激样品进行还原、烷基化并用胰蛋白酶进行酶切。

Byos 包括 SVA 和 PTM 分析的workflow,并且这些workflow已经包含序列变异体分析和翻译后修饰的典型修改。
以 Byos 工作流程为基础,用户可以根据不同的需求(例如不同的仪器配置或理论的修饰类型)进行自定义设置。SVA 工作流程预先配置了单氨基酸突变/错误翻译的所有预期序列变异体。如果需要,也可以轻松添加其他修饰。
高级配置可用于启用验证器,分别如图 1A 和 1B 所示的序列变异体验证器和修饰验证器。然后,验证器算法将在 Byos 的“Comments”列中添加“Critical”、“Warning”或“Clear”标签,以描述严重性以及错误识别的原因。如果使用默认参数,则无需添加 [SVAOptions] 或 [ModValidatorOptions] 高级配置命令。如果用户想要更改默认参数,可以相应地添加和修改验证器选项命令。
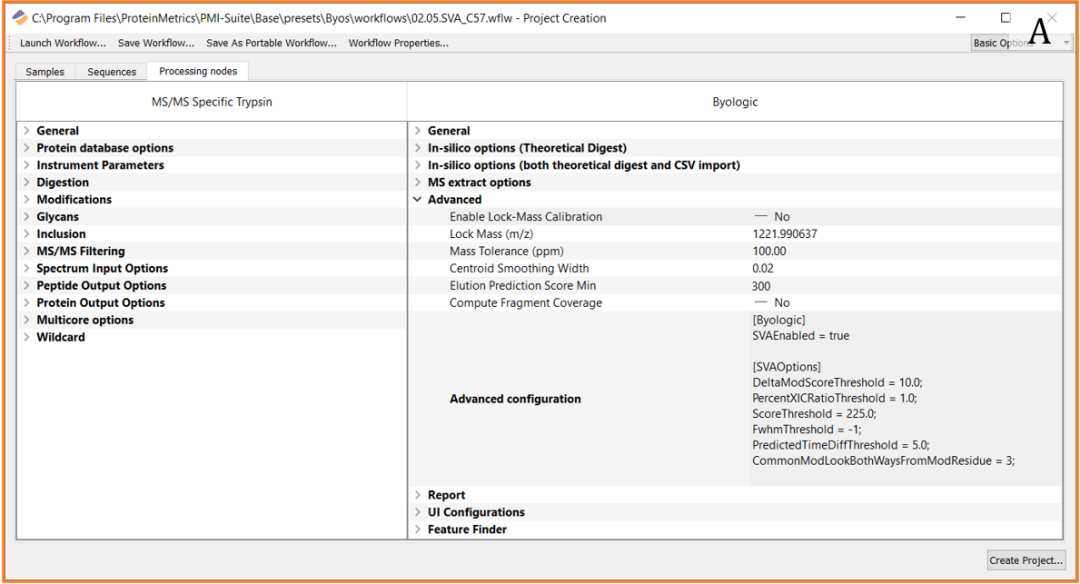
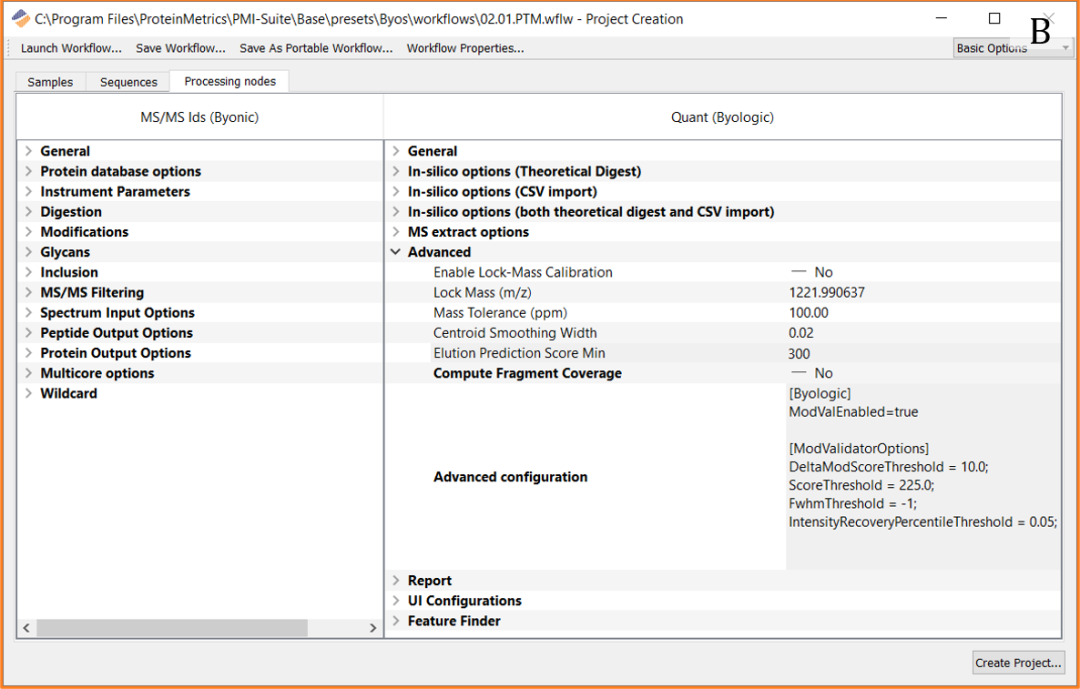
图 1. Byos 项目创建窗口。A:显示已启用序列变异体验证器的高级配置。B:显示已启用修饰验证器的高级配置。对于序列变异体验证器和修饰验证器,第一个命令便是打开验证器所需的所有命令。如果用户想要更改任何默认参数,[SVAOptions] 或 [ModValidatorOptions] 可以根据需要添加和修改。
结果与讨论
序列变异体分析
• 使用 Byos 序列变异体验证器,可以按照图 2 所示的逻辑简化排除误报和验证真实标识的过程。
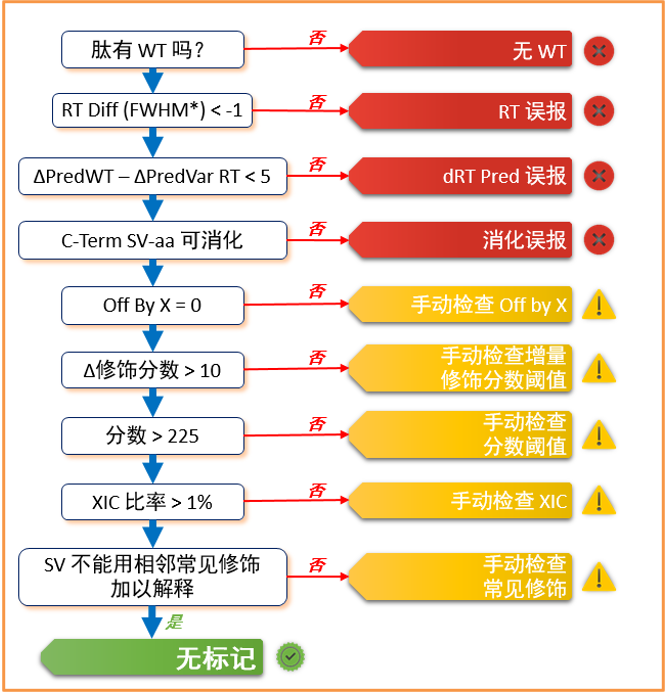
图 2. 当验证器算法部署在 Byos 中时,用于标记序列变异体的逻辑。带有“Critical”标签的序列变异体很可能是误报。带有“Warning”标签的序列变异体应进行手动检查。带有“Clear”标签的序列变异体很可能是真实的阳性结果,并且不需要大量的时间进行审查。RT Diff、ΔPredWT-ΔPredVarRT、ΔModScore, Score和XIC Ratio %的默认阈值如上所示。通过更改高级命令中的值,可以根据分析人员的偏好更改这些阈值。
*注:FWHM 作为最小保留时间差,将修饰分类为不与其野生型共洗脱。设置为 -1 可确定输入文件的平均 FWHM,随后自动计算阈值。或者,可以使用任何大于 0 的实数进行手动设置。
• 使用验证器功能来评估 NIST mAb 的对照和应激样品中序列变异测定的准确性,数据带有为“Critical”、“Warning”和“Clear”标记。
• 使用这些标记,可以快速审查数据并将其标记为误报或真实标识。
• 图 3 显示了项目中生成的标签示例。数据经过筛选后仅显示序列变异体及其相应的未修饰肽段(“野生型”)
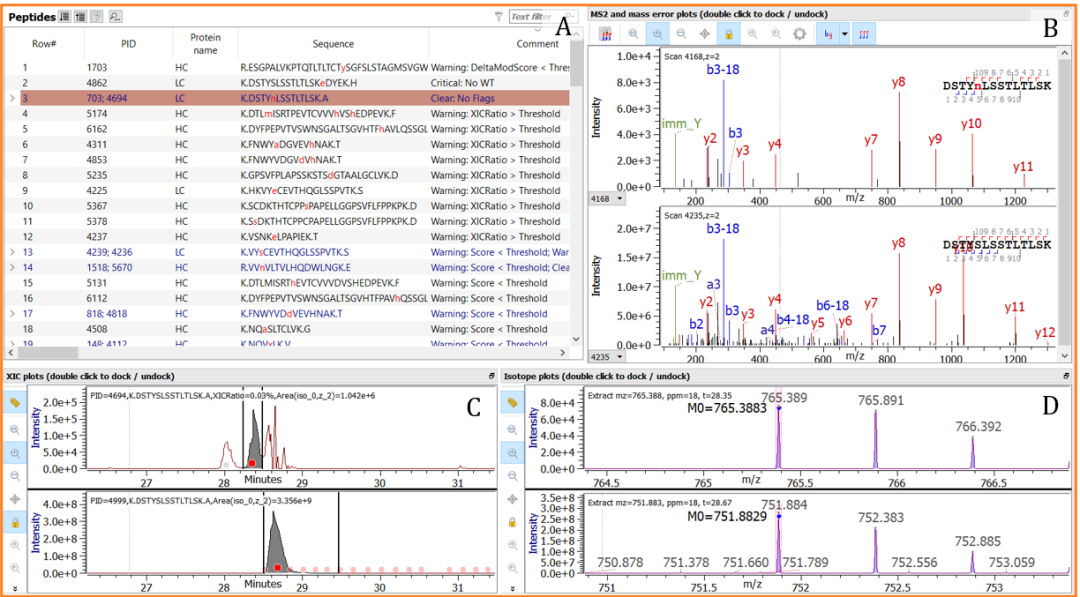
图 3:使用默认 Byos 工作流程和通过高级配置选项启用的验证器进行 SVA 的结果。该图表只显示了选择的肽段的相关信息 (A)、所选肽及其野生型的 MS/MS 谱图 (B)、提取离子色谱图 (C) 和 MS 谱图 (D)。每个序列变异体都会在“Comment”列 (A) 中自动标记为“Critical”、“Warning”或“Clear”,以实现快速数据审查并轻松筛选报告数据。此处选择的肽段标签显示 “Clear:No Flags”,由此很容易看出这是一个真正的序列变异体。MS 谱图 (D) 显示了丝氨酸-天冬酰胺置换的预期质量转移(2+ 离子为 13.5)。MS/MS 谱图清楚地显示了 y 离子系列中 y10 处 (B) 变异体和野生型之间的质量迁移,并且变异体的保留时间存在差异 (C)。此外,从这些数据可以看出,很明显该变异体的存在水平比野生型低得多,这符合预期。
• 有许多已知的机制可以在序列变异体分析中轻松标记误报结果。这些问题会生成“Critical”标签,具体如图 2 红色部分所示。
• 无野生型:在受控蛋白质生成中,序列变异体只会出现在产品的很小一部分中。因此,预计会检测到未置换的肽段序列(“野生型”)。如果没有证据表明存在野生型,那么所提出的变异体将被视为错误分配。
• Off by X=0:允许对前体同位素偏移进行错误检查。在许多仪器上,标称前体质量实际上可能是 13C 同位素峰的质量,而不是基峰(所有 12C 单同位素)的质量。
• RT Diff < 0.2:由于两种肽之间的氨基酸差异,通常会通过色谱法分离真正的序列变异体及其未修饰的肽序列。野生型肽和检测到的变异体保留时间没有差异,这是 SVA 中误报的自动标记。最小保留时间差的默认设置为 0.2,但用户可以根据需要进行修改。
• ΔPredWT – ΔPredVar RT < 5:软件可以预测肽段相对于其他肽段(例如野生型“WT”)的洗脱位置。如果此预测明显偏离,则会在“Comment”列中返回严重警告。
• 图 4 显示了“RT Diff < 0.2”的示例。
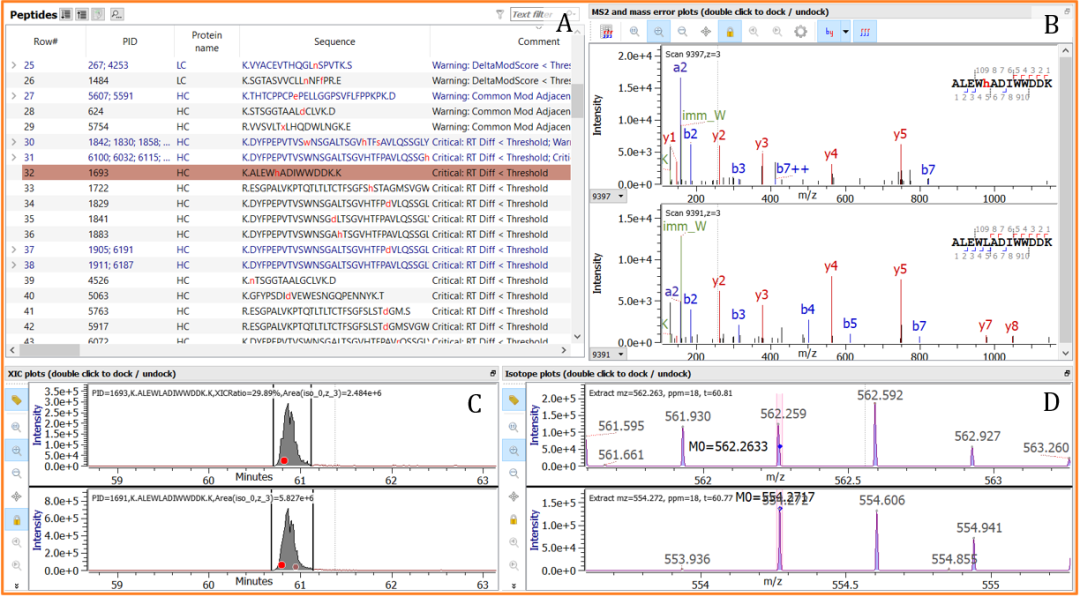
图 4:使用默认 Byos 工作流程和通过高级配置选项启用的验证器进行 SVA 的结果。屏幕截图只显示了选择的肽段的相关信息(A)、所选肽及其野生型的 MS/MS 谱图 (B)、提取离子色谱图 (C) 和 MS 谱图 (D)。每个序列变异体都会在“Comment”列 (A) 中自动标记为“Critical”、“Warning”或“Clear”,以实现快速数据审查并轻松筛选报告数据。此处选择的肽段标签显示“Critical: RT Diff < Threshold”,由此很容易确定这是一个误报。首先,如标签所示,所提出的变异体和野生型的色谱图谱是相同的 (C),这对于氨基酸置换来说是非常不寻常的。MS 谱图 (D) 显示单同位素质量(由蓝点表示)较之变异体的峰值小 1 Da,从而表明出现错配。MS/MS 谱图 (B) 显示序列离子之间的差异非常小,并且没有证据表明 y 离子高于 y9。指示变异体任何可能性的 b 离子 (b7) 丰度极低。此外,从这些数据可以看出,很明显所提出的变异体与野生型的数量级相同,这进一步表明出现了错配。
• 使用验证器算法和注释列,可以快速审查数据,从而使用筛选选项消除误报。
翻译后修饰分析
• 使用 Byos 修饰验证器,可以按照图 5 所示的逻辑简化排除假标识和验证真实标识的过程。
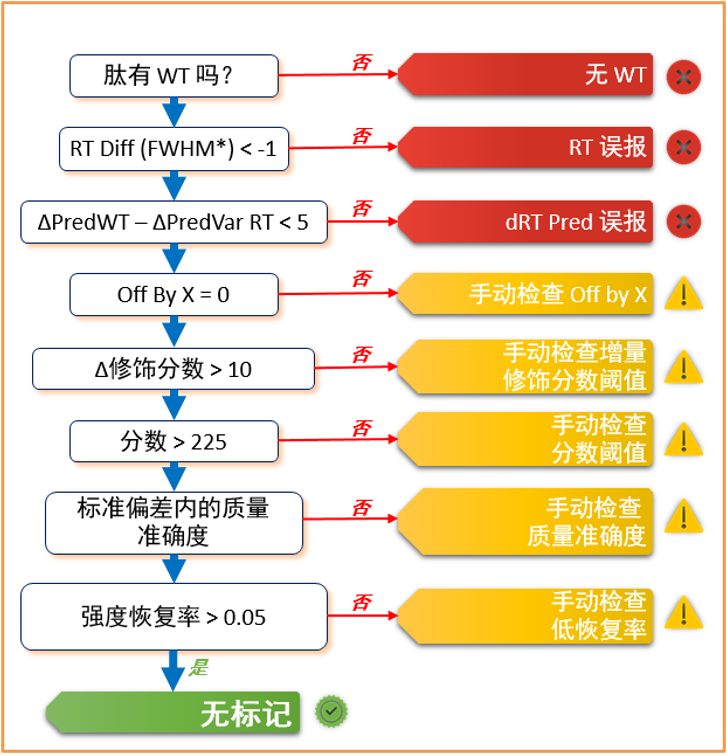
图 5. 当修饰算法部署在 Byos 中时,用于标记修饰的逻辑。带有“Critical”标签的修饰很可能是误报。带有“Warning”标签的序列变异体应进行手动检查。带有“Clear”标签的序列变异体很可能是真实的阳性结果,并且不需要大量的时间进行审查。
*注:FWHM 作为最小保留时间差,将修饰分类为不与其野生型共洗脱。设置为 -1 可确定输入文件的平均 FWHM,随后自动计算阈值。或者,可以使用任何大于 0 的实数进行手动设置。
• 在与序列变异体分析相同的项目中,验证器功能用于标记 NIST mAb 对照样品和应激样品中的修饰肽段。
• 使用数据标签“Critical”、“Warning”和“Clear”,可以快速审查数据并将其标记为误报或真实标识。
• 图 6 显示了项目中生成的修饰验证器标签的示例。
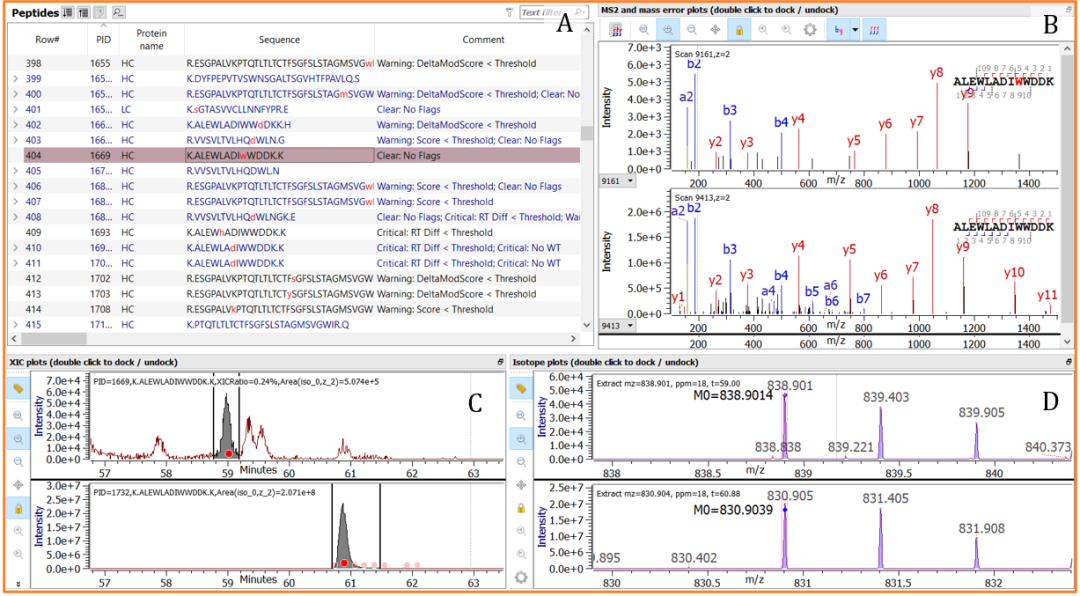
图 6:使用默认 Byos SVA 工作流程和通过高级配置选项启用的验证器进行 PTM 分析的结果。屏幕截图只显示了选择的肽段的相关信息(A)、所选肽及其野生型的 MS/MS 谱图 (B)、色谱图 (C) 和 MS 谱图 (D)。每个修饰的肽都会在注释列 (A) 中自动标记为“Critical”、“Warning”或“Clear”,以实现快速数据审查并轻松筛选报告数据。此处选择的肽段标签显示“Clear: No Flags”,由此可以看出这是真正的色氨酸氧化。MS 谱图 (D) 显示了预期的氧化质量转移(2+ 离子增加 8 Da)。MS/MS 谱图中的 y 离子系列(图 7 中放大)清楚地显示了氧化肽和野生型 (B) 之间的质量转移,并且正如预期的那样,氧化肽洗脱远远早于野生型 (C)。此示例显示标签为“Clear: No Flags”,表示数据不需要进行人工审查,可以标记为真实结果。
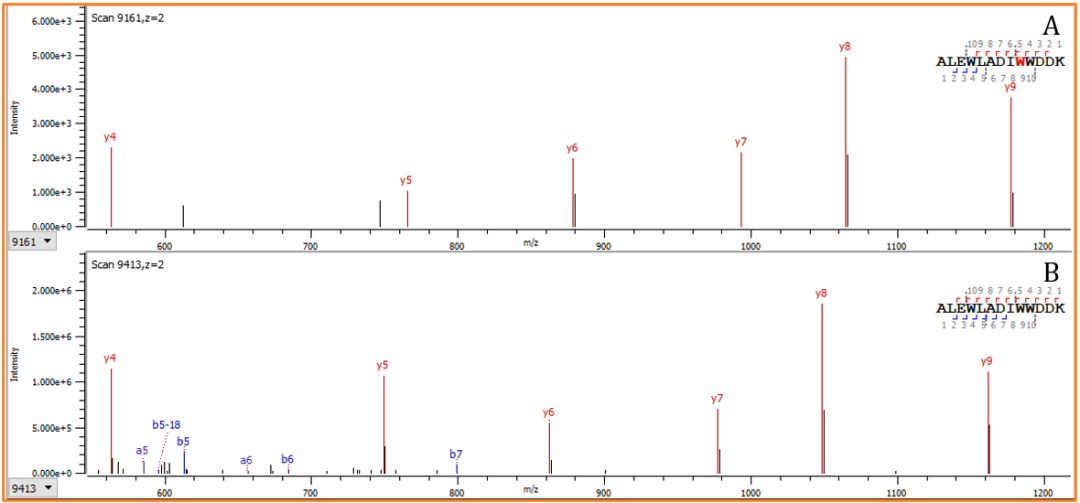
图 7:图 6 中所选肽的 MS/MS 谱图。y 离子系列(从 y5 到 y9)清楚地显示了氧化肽 (A) 和野生型 (B) 之间的质量转移。
• 与 SVA 一样,有许多已知的机制可以在 PTM 分析中轻松标记误报。这些问题会生成“Critical”标签,具体如图 5 所示。
• 图 8 显示了“Critical: RT Diff < Threshold”标记。从所提出的修饰肽段及其未修饰对应物的色谱图中可以清楚地看出,两者之间没有保留时间差。
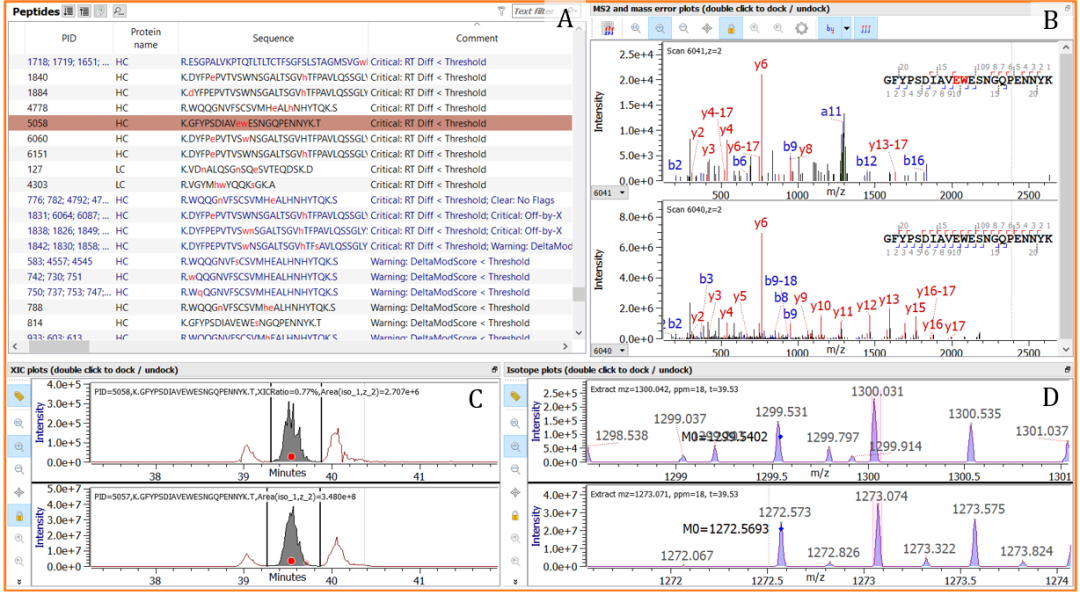
图 8:使用默认 Byos SVA 工作流程和通过高级配置选项启用的验证器进行 PTM 分析的结果。屏幕截图只显示了选择的肽段的相关信息(A)、所选肽及其野生型的 MS/MS 谱图 (B)、提取离子色谱图 (C) 和 MS 谱图 (D)。这里所提出的氧化肽可以立即被视为错误分配而被驳回,因为它显示标签“Critical: RT Diff < Threshold”。如标签所示,所提出的修饰肽和野生型的色谱图相同 (C),这表明如果肽段上存在氧化,则它发生在 MS 源中。数据不需要进一步询问,但如果询问的话,没有强有力的支持证据来支持氧化形式。
• 验证器算法自动填充到“Comment”列中的文本极大地简化了数据审查流程,并将审查者引导至需要用户验证的标识。
• 当使用验证器注释审查数据时,序列变异体或修饰可以在 Validate 列中标记为 True-positive、False-positive 或 Uncertain,具体如图 9 所示。
• 在数据审查期间使用“Validate”列表,使用户能够清楚验证数据是否已经过手动检查。Validate 列用于筛选报告,仅查找已由分析人员验证的真实结果。
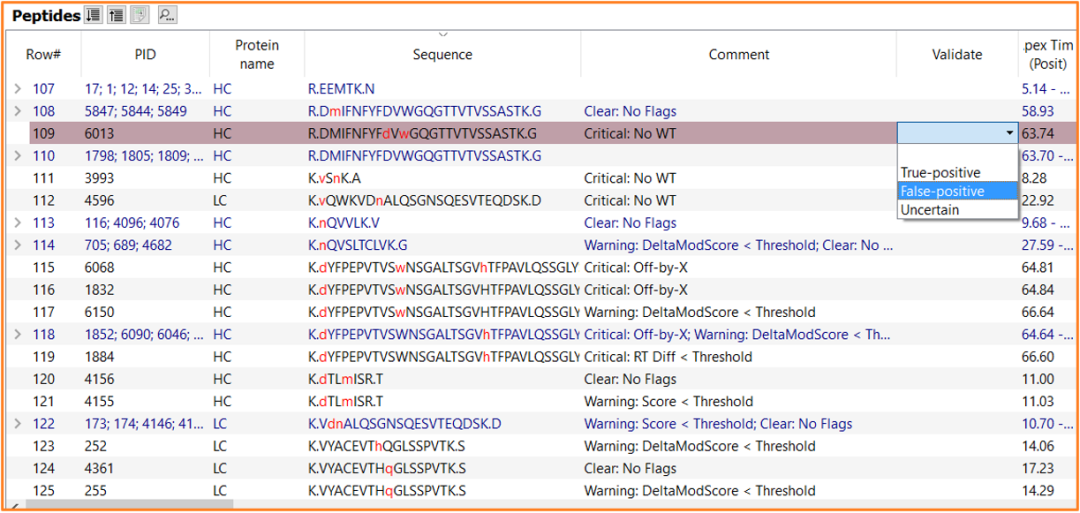
图 9:使用默认 Byos SVA 工作流程和通过高级配置选项启用的验证器进行 PTM 分析的结果。屏幕截图显示了选择一种肽的肽表。“Validate”列可用于将每个识别标记为“True-positive”、“False-positive”或“Uncertain”。
结论
尽管数据审查对于绝大多数通过 LC-MS/MS 进行的生物治疗药物分析来说非常必要,但 Byos 中的自动化工具明显有助于简化流程。通过在 Byos 的高级配置中启用修饰验证器和序列变异体验证器,审查者可以使用自动生成的注释来快速排除误报并列出真实结果。之后,可以对需要进一步彻底审查的数据进行适当分类和一致性评估,无需考虑时间或者分析人员、仪器或样品制备方面的差异。
有关翻译后修饰和序列变异体分析工作流程的更多资源和背景信息,请访问 https://www.proteinmetrics.com/workflows/ 或通过 support@proteinmetrics.com 联系客户成功团队。
关于Protein Metrics
Protein Metrics LLC是一家全球领先的质谱数据解析软件供应商,公司总部位于美国加州。我们为科研和企业用户提供高效准确的一站式质谱数据解析方案,帮助用户发现、解决问题。PMI 在全球范围内提供销售和支持,目前已为超过150个企业和300个科研单位提供服务。
联系我们邀约演示:
王蕾 13482181958
Protein Metrics Inc.
Cupertino, California
USA
www.proteinmetrics.com
info@proteinmetrics.com


